《表2 脱靶序列预测信息:基于优化sgRNA系统提高海岛棉CRISPR/Cas9基因组编辑功效的研究》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于优化sgRNA系统提高海岛棉CRISPR/Cas9基因组编辑功效的研究》
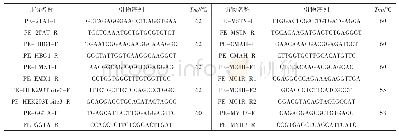
加粗部分为靶序列PAM位点。Bold part shows the target sequence PAM sites.
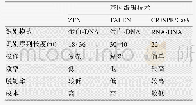
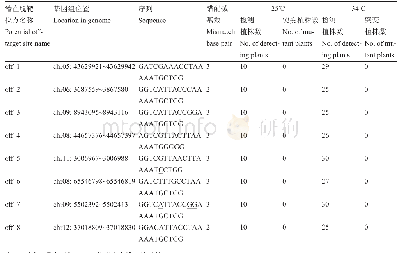
根据设计的Guide RNA序列,浏览华中农业大学CRISPR sgRNA设计网站(http://crispr.hzau.edu.cn/cgi-bin/CRISPR2/CRISPR)[6],寻找本研究设计使用的GGB-sgRNA1和Shift GGB-sgRNA1靶序列,并分别预测得到脱靶率较高的2种靶序列(表2),利用该序列,登录中国农业科学院棉花基因组数据库(https://cgp.genomics.org.cn/),通过BLAST获得每条预测的脱靶序列对应的同源序列,再通过Mapping View将上述核苷酸序列定位到具体的染色体进而获得涵盖该脱靶序列的基因组序列,设计用于检测脱靶率的引物(表3)。
| 图表编号 | XD0096084100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.10.12 |
| 作者 | 李继洋、胡燕、姚瑞、代培红、刘晓东 |
| 绘制单位 | 新疆农业大学农学院、新疆农业大学农业生物技术重点实验室、石河子大学生命科学院、新疆农业大学农学院、新疆农业大学农业生物技术重点实验室、石河子大学生命科学院、新疆农业大学农学院、新疆农业大学农业生物技术重点实验室、新疆农业大学农学院、新疆农业大学农业生物技术重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |