《表1 本研究所用ITS序列的物种名、标本号和序列号》
注:本研究新测序的序列GenBank号以粗体显示
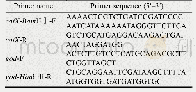
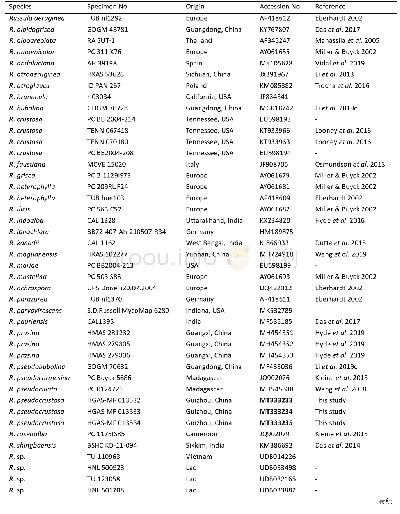
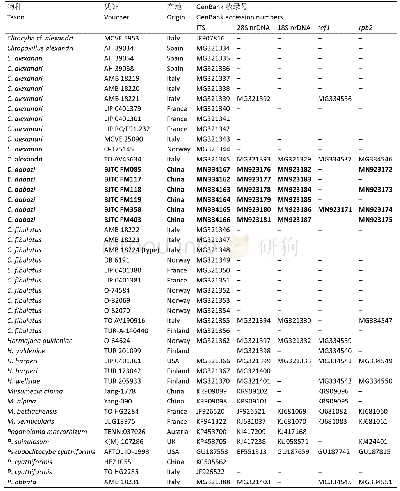
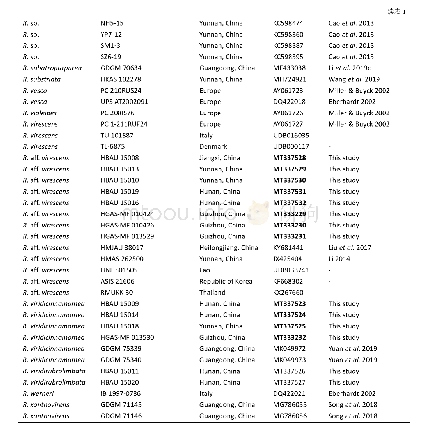
DNA提取采用改良CTAB法,基因组DNA和PCR产物的检测均采用1%的琼脂糖凝胶电泳法(李国杰2014)。ITS序列扩增和测序引物为ITS1和ITS4(White et al.1990),扩增产物经紫外光检测后,切胶纯化回收并送往北京博迈德基因技术有限公司进行双向测序,所得序列用Chromas v2.6.6软件检测峰图质量(http://technelysium.com.au/wp/chromas),使用DNASTAR Lasergene v11.1软件包中的SeqMan组件拼接获得高质量的完整序列(Allex 1999),并上传至G e n B a n k数据库(M T 3 3 3 2 2 9–M T 3 3 3 2 3 5,MT337523–MT337532)。利用所测序列,在GenBank和UNITE数据库中检索获取相似性较高的同源序列。将本研究所测序列、亚组内近似种类序列和组内其他分类群代表序列汇总(表1),利用Mafft v7.450软件对序列进行比对(Katoh et al.2013),通过BioEdit v7.2.5软件对比对结果进行手动调整(Hall 1999)。最大似然法(maximum likelihood,ML)系统发育分析使用RaxML v7.2.6软件(Silvestro&Michalak 2012;Stamatakis2014),碱基替换模型选择GTRGAMMA,经1 000次非参数自助法(nonparametric bootstrapping)分析获得支持度。贝叶斯似然法(Bayes)系统发育分析使用Mr Bayes v3.2.7软件(Ronquist et al.2012),核酸替代模型筛选使用MrModeltest v2.3程序在PAUP v4.0b10软件中进行。异褶组Russula sect.Heterophyllae和变绿亚组Russula subsect.Virescentinae系统发育分析选用的核酸替代模型分别为HKY+I+G和HKY+G。系统发育树使用Fig tree v1.4.4和Adobe Illustrator CS6进行可视化和编辑。
| 图表编号 | XD00222836000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.22 |
| 作者 | 邓春英、史路瑶、王晶、向准、李守勉、李国杰、杨慧 |
| 绘制单位 | 贵州省生物研究所贵州科学院、河北农业大学园艺学院河北省种质资源创新利用重点实验室河北省蔬菜产业协同创新中心、贵州省生物研究所贵州科学院、贵州省生物研究所贵州科学院、河北农业大学园艺学院河北省种质资源创新利用重点实验室河北省蔬菜产业协同创新中心、河北农业大学园艺学院河北省种质资源创新利用重点实验室河北省蔬菜产业协同创新中心、食用菌工程中心铜仁职业技术学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式