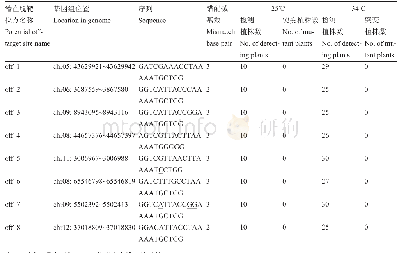
《表2 CRISPR/Cas9潜在脱靶位点的突变检测》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《利用CRISPR/Cas9技术创制水稻落粒性基因qSH1突变体及突变特征分析》
加粗字母为与sgRNA匹配的碱基;标注下划线字母为PAM区序列The base that matched the sgRNA are marked in bold;The sequences of the PAM are underlined
为检测各个靶位点的突变情况,跨靶点T1设计特异性靶点检测引物qSH1-JC-F2/qSH1-JC-R2(表1),目的片段的大小约为772 bp,T2、T3两个靶位点检测引物与T1靶点检测引物相同;跨Ta、Tb两个靶位点设计特异性靶点检测引物qSH1-JC-F1/qSH1-JC-R1(表1),目的片段的大小分别约为671bp。对T0代转基因阳性植株利用相对应的特异性靶点检测引物进行PCR扩增(反应程序及条件参照1.3中质粒DNA扩增),胶回收目的片段,送湖南擎科生物测序。利用MAGE4.0分析测序结果,在靶点位置出现与HR1128野生型序列有差异的单峰或双峰、套峰的单株即为T0代突变植株。之后将突变植株扩增的目的片段进行TA克隆,挑选10个阳性单克隆进行测序,将测序结果与HR1128野生型序列进行对比分析靶位点的突变类型及基因型。利用NCBI、Gramene的Blast工具,选择Oryza sativa Indica Group为参考序列,筛选与每个sgRNA序列匹配度大于或等于15 bp且3'端具有NGG结构的20 bp片段作为潜在脱靶位点(表2),在不同类型的T0代转基因阳性植株中对相应的潜在脱靶位点进行扩增、测序检测(脱靶位点扩增检测体系、程序与qSH1-JC-F2/R2一致),以评估各个靶位点的脱靶效应。
| 图表编号 | XD0038774300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.02.25 |
| 作者 | 盛夏冰、汪雪峰、谭炎宁、孙志忠、余东、袁贵龙、袁定阳、段美娟 |
| 绘制单位 | 湖南农业大学生物科学技术学院、湖南杂交水稻研究中心、湖南农业大学生物科学技术学院、湖南杂交水稻研究中心、湖南杂交水稻研究中心、湖南杂交水稻研究中心、湖南农业大学生物科学技术学院、湖南杂交水稻研究中心、湖南杂交水稻研究中心、湖南杂交水稻研究中心、湖南农业大学生物科学技术学院 |
| 更多格式 | 高清、无水印(增值服务) |