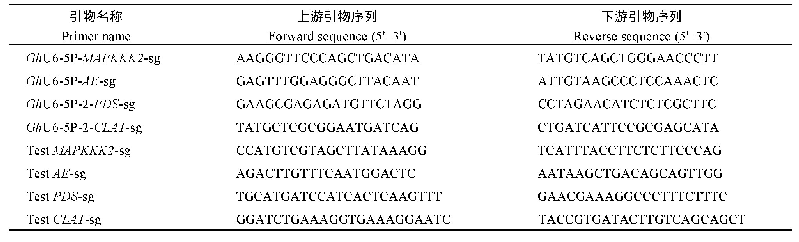
《表1 本研究中使用的序列信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《云南西部克钦湍蛙的鉴定与描述及克钦湍蛙在中国的分布讨论》
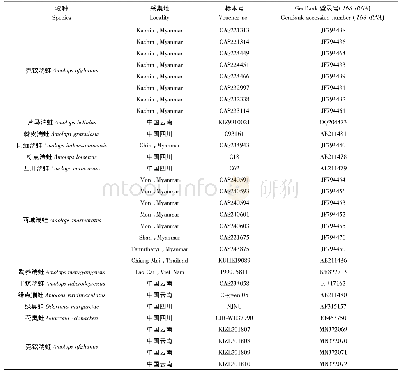
首先使用lasergene中的sequman对原始序列进行剪切,得到长度为850 bp的片段。本实验扩增了克钦湍蛙序列,并从NCBI网站下载了部分湍蛙属物种的16S rRNA序列以及花臭蛙Odorrona schmackeri和绿臭蛙Odorrona margaretae的16S rRNA序列作为外群(表1)。使用Clustal X1.83(Thompson et al.,1997)进行序列比对。物种间的遗传距离(P-distances)使用MEGA 5计算(Tamura et al.,2011)。在MEGA 7 (Kumar et al.,2016)中筛选最适合该数据集序列演化模型进行最大似然法(Maximum Likelihood,ML)分析和贝叶斯分析(Bayesian Inference,BI)。根据BIC标准,适合本数据集的模型为GTR+G。使用MEGA 7构建最大似然树,系统进化树的测试方法为Bootstrap,重复次数设为1 000,进化距离模型选择General Time Reversible Model,Rates among sites设置为Gamma Distributed (G),分支交换算法设置为SPR level 3。使用Mr Bayes 3.2 (Ronquist et al.,2012)构建BI树,替换模型设置为GTR模型,位点间差异比率设为gamma,使用马尔可夫链-蒙特卡罗(MCMC)数据模拟技术来估算后验概率,运算2 000 000代,隔1 000代抽样一次(王勇等,2009),并通过设置参数“diagnosis”来计算2个随机起始程序的平均标准差,burn-in即为标准差在0.01以前的抽样数,BI树即根据剩余样本构建的50%一致树(consensus tree)。BI树上各分支节点的支持率以后验概率(posterior probability,PP)表示。
| 图表编号 | XD00224846600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.28 |
| 作者 | 刘硕、饶定齐 |
| 绘制单位 | 中国科学院昆明动物研究所昆明动物博物馆、中国科学院昆明动物研究所 |
| 更多格式 | 高清、无水印(增值服务) |