《表3 茶树不同生育期腋芽转录组数据质量报告》
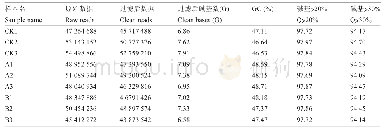
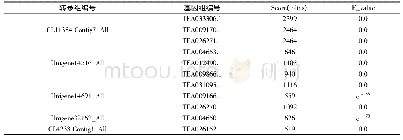
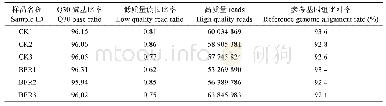
我们分析了不同生育期腋芽的全转录组数据,数据质量报告见表3。通过比较各生育期的基因表达情况,一共筛选出961条差异表达基因(P<0.05)。将这些差异表达基因与拟南芥的基因组进行比对,在茶树基因组数据库(http://tpia.teaplant.org)上进行基因功能查询,结果发现有8个与碳氮代谢相关的基因表达差异显著(7个编码ATP酶家族基因:TEA005087、TEA006851、TEA014096、TEA016662、TEA016722、TEA025126、TEA029704,1个编码SPS酶基因:TEA030791)(表4)。在本研究中,我们发现基因TEA029704在休眠期时高表达,随着发育的进程,表达量逐渐降低。基因TEA029704与AT3G21180相似性高达93.103%,KEGG通路分析其为编码Ca2+-ATP酶的基因;TEA016722与TEA006851在休眠期低表达,在1芽1叶期高表达。基因TEA016722与AT2G34660相似性为59%,KEGG通路分析为编码热激蛋白基因;基因TEA006851随着发育的进程,表达逐渐降低,与AT3G12580相似性为93.739%,KEGG通路分析显示其为编码ATP依赖的蛋白水解酶基因。基因TEA030791与AT1G04920相似性达73.166%,KEGG通路分析显示其为编码蔗糖磷酸合成酶基因。
| 图表编号 | XD00100024000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.07.30 |
| 作者 | 吴琼、刘丹丹、阮旭、沈季雪、徐奕鼎、王文杰、张正竹、夏恩华 |
| 绘制单位 | 安徽省农业科学院茶叶研究所、安徽农业大学省部共建茶树生物学与资源利用国家重点实验室、安徽省农业科学院茶叶研究所、安徽省农业科学院茶叶研究所、安徽省农业科学院茶叶研究所、安徽省农业科学院茶叶研究所、安徽省农业科学院茶叶研究所、安徽农业大学省部共建茶树生物学与资源利用国家重点实验室、安徽农业大学省部共建茶树生物学与资源利用国家重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式