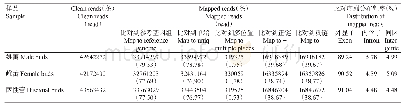
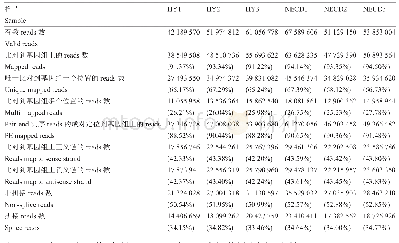
《表2 样品和参考基因组比对情况》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于RNA-Seq技术的葡萄不同花型新转录本预测和基因结构优化》
我们对RNA-Seq测序的原始数据进行质量控制。首先去除了原始数据中的接头污染,并且对含有未知碱基比例大于5%的片段和质量值小于20的片段进行了过滤,最终得到了173.41 Gb干净数据(clean data)。利用TopHat2软件(v2.0.12版),设置参数mismatch=2,对各样品中过滤后的测序序列进行基因组定位分析。各样品的clean reads与参考基因组PN40024的序列比对效率为56.71%~69.65%,各样品Q30[质量值(Q)越高代表碱基被测错的概率(P)越小,其计算公式为Q=-10lg P,Q30代表碱基被测错的概率为1‰]碱基百分比均≥88.74%,表明碱基识别准确度较高。如表2所示,各样品比对到指定参考基因组上的匹配读段均不低于总数的56.71%。其中,比对到参考基因组唯一位置的单一匹配读段(uniquely mapped reads)均不低于55.45%;比对到参考基因组多处位置的多匹配读段(multiply mapped reads)平均为1.27%,这些读段对应的基因可能是多拷贝基因。碱基类型分布情况检查表明,测序数据无AT、GC分离现象,且GC含量平均在46.56%左右(表2)。
| 图表编号 | XD0086762100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.05.20 |
| 作者 | 纪薇、焦晓博、罗尧幸、赵伟、郭雯岩、刘榕晨、赵旗峰、马小河 |
| 绘制单位 | 山西农业大学园艺学院、果树种质创制和利用山西省重点实验室、农业农村部黄土高原作物基因资源与种质创制重点实验室、山西农业大学园艺学院、果树种质创制和利用山西省重点实验室、农业农村部黄土高原作物基因资源与种质创制重点实验室、山西农业大学园艺学院、果树种质创制和利用山西省重点实验室、农业农村部黄土高原作物基因资源与种质创制重点实验室、山西农业大学园艺学院、果树种质创制和利用山西省重点实验室、农业农村部黄土高原作物基因资源与种质创制重点实验室、山西农业大学园艺学院、果树种质创制和利用山西省重点实验室、农业农村部黄 |
| 更多格式 | 高清、无水印(增值服务) |