《表1 RNA-Seq数据的比对统计》
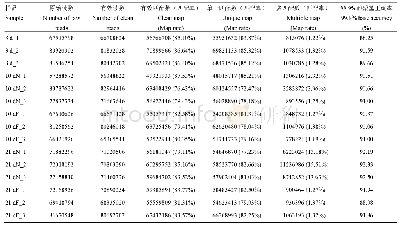
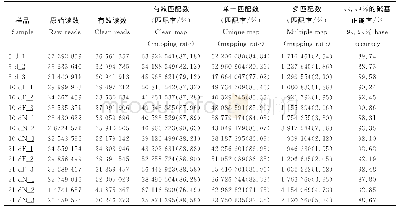
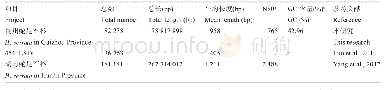
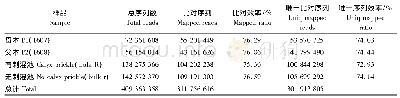
基于边合成边测序(Sequencing By Synthesis,SBS)技术,利用IlluminaHiSeq4000高通量测序平台对双亲、混池的4个cDNA文库进行测序,共获得60.77 Gb的Clean Data,各样品Q30碱基百分比在88.9%以上(表1)。经过原始数据处理,4个文库共获得409 363 358个Clean Reads,利用Bowtie 2软件将Clean reads与参考基因组进行比对,共有311 756 616个比对到基因组,比对效率均大于75.00%。有刺混池和无刺混池分别有104 202 238个和109 191 303个Clean reads比对到参考基因组,其中比对到唯一的位置的Uniq Mapped Reads分别为72.93%和74.14%。以上比对结果表明,从建库到测序等均达到试验要求。
| 图表编号 | XD0063698700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.08.28 |
| 作者 | 潜宗伟、陈海丽、崔彦玲 |
| 绘制单位 | 北京市农林科学院蔬菜研究中心农业部华北地区园艺作物生物学与种质创制重点实验室蔬菜种质改良北京市重点实验室、北京市农林科学院蔬菜研究中心农业部华北地区园艺作物生物学与种质创制重点实验室蔬菜种质改良北京市重点实验室、北京市农林科学院蔬菜研究中心农业部华北地区园艺作物生物学与种质创制重点实验室蔬菜种质改良北京市重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式