《表4 Illumina测序数据统计分析》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《鳗鲡养殖废弃物抗性基因赋存特征及其与抗生素和微生物群落的相关性》
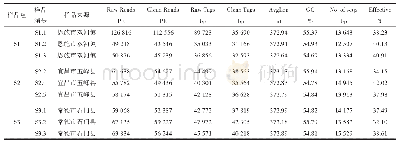
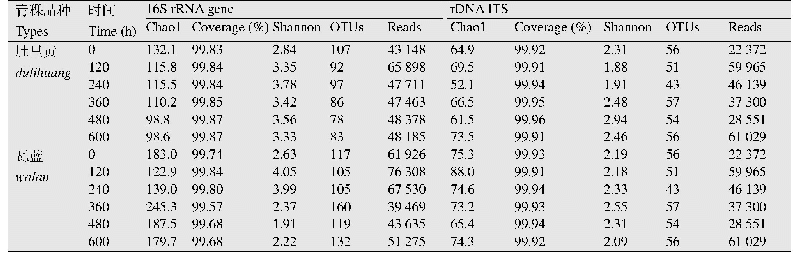
采用IlluminaNovaSeq高通量测序对鳗鲡养殖废弃物样品中的微生物菌群多样性和组成结构进行分析,8个鳗鲡养殖废弃物样品获得的有效16S rRNA序列数为82 131-99 246条,共计608 526条.基于97%的序列相似性,各样品获得的OTUs数为54 465-67 172.如表4所示,Good’s coverage指数在99%以上,表明本研究获取的数据量能够很好地反映样品的微生物群落多样性情况.Alpha多样性分析结果表明,底泥和废水中微生物群落的物种丰度指数(observed species,ACE和Chaol)和多样性指数(Shannon和Simpson)都存在一定的差异.除MLS3和MLW3外,底泥的细菌物种数和群落多样性都要高于养殖废水中的微生物群落.根据weighted Unifrac计算beta多样性距离矩阵,基于距离矩阵进行主坐标分析(principal coordinates analysis,PCoA),样本间的距离越大,说明样本间的细菌群落差异越大.PCoA分析结果(图3)表明,鳗鲡养殖废弃物样品可以根据环境介质进行聚类分组,相同环境介质样品在微生物结构和组成方面是相近的.
| 图表编号 | XD00200349200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.10.25 |
| 作者 | 黄薇、刘兰英、刘洋、罗土炎、宋永康 |
| 绘制单位 | 福建省农业科学院农业质量标准与检测技术研究所、福建省农产品质量安全重点实验室、福建省农业科学院农业质量标准与检测技术研究所、福建省农产品质量安全重点实验室、福建省农业科学院农业质量标准与检测技术研究所、福建省农产品质量安全重点实验室、福建省农业科学院农业质量标准与检测技术研究所、福建省农产品质量安全重点实验室、福建省农业科学院农业质量标准与检测技术研究所、福建省农产品质量安全重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |