《表4.突变酶的酶活测定:Pseudomonas putida KT2440低特异性L-苏氨酸醛缩酶的表达、酶学性质及温度稳定性提高》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《Pseudomonas putida KT2440低特异性L-苏氨酸醛缩酶的表达、酶学性质及温度稳定性提高》
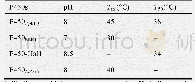
对不同来源的苏氨酸醛缩酶的氨基酸序列和二级结构进行比对分析(图7),发现Thr206在嗜热性菌存在,很可能与酶的温度稳定性相关,而Lys207位于保守区域,并与催化中心相邻,因而选择这两个位点进行突变。通过全质粒PCR引入突变T206S、K207A、K207R、K207S和K207T,这些突变不影响蛋白在大肠杆菌中的表达。酶活测定结果表明Lys207突变酶对L-苏氨酸裂解活性丧失或者几近失活,酶活仅为野生型的0.3%–4.8%(表4),说明Lys207对酶活性有很大的影响。通过对与LTA相似性为43.14%的苯基丝氨酸醛缩酶的晶体结构(PDB ID:1V72)分析表明,Lys207能够与磷酸吡哆醛(PLP)结合形成希夫碱(图8),其催化机制为辅因子PLP与赖氨酸的ε-氨基形成内部醛亚胺,在苏氨酸的进攻下,PLP和苏氨酸的α-氨基形成外部醛亚胺,随后裂解产生乙醛和PLP-甘氨酸醌类复合物,甘氨酸的α-碳原子质子化后,释放出甘氨酸,并且PLP与活性位点赖氨酸残基再生内部醛亚胺。赖氨酸突变成其他的氨基酸后导致不能与PLP结合,使得酶的催化效率大大降低甚至没有活性[21]。而T206S对酶活大小几乎没有影响(表4)。圆二色谱可以用于探究蛋白质的热变性曲线,随着温度的升高,蛋白质内部的α-螺旋会逐渐解螺旋,根据蛋白质的解螺旋速度可以作出变性曲线,从而确定蛋白的变性温度,即Tm值,蛋白变性温度是蛋白质的二级结构解螺旋速率达到最大时所对应的温度,即变性曲线最大斜率处所对应的温度。通过计算曲线的最大斜率,KT2440野生型的变性温度为49.2?C,而T206S突变酶的变性温度为53.7?C,相比于野生型LTA酶,变性温度提高了4.5?C,说明该位点与酶的温度稳定性密切相关(图9)。
| 图表编号 | XD0095874300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.10.04 |
| 作者 | 李利宏、张荣珍、周丽仙、柳志永、旋凯昂、饶俊超、徐岩 |
| 绘制单位 | 江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室、江南大学生物工程学院教育部工业生物技术重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |