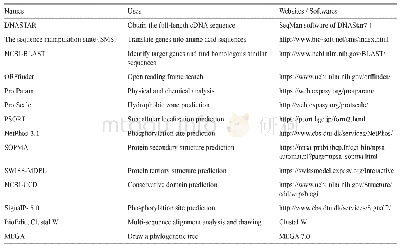
《表2 生物信息学分析网站》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《鹅掌楸LcKNOX3转录因子基因全长克隆及组织表达特异性分析》
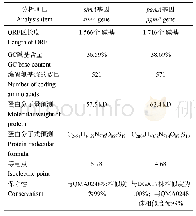
在NCBI ORF finder网站上在线预测LcKNOX3基因的开放阅读框,并翻译成氨基酸序列。将得到的开放阅读框(ORF)序列通过TAIR拟南芥资源信息网站上与拟南芥基因进行比对,在TAIR上下载拟南芥有报道的7个KNOX基因家族编码的氨基酸序列,使用DNAMAN软件进行序列比对。在NCBI conserved domain中预测LcKNOX3蛋白的保守结构域,利用Protparam对LcKNOX3蛋白一级理化性质进行预测,根据predict protein在线预测LcKNOX3编码各种氨基酸所占比例。采用SignalP程序对LcKNOX3基因进行蛋白质信号肽分析。通过登录SOPMA网站预测分析LcKNOX3蛋白的二级结构;使用Phyre2工具进一步预测蛋白三级结构并建模。将LcKNOX3基因编码氨基酸序列在NCBI数据库中进行blastp比对,下载blast分值较高的几个物种的氨基酸序列,利用MEGA 6.0进行多序列比对(Tamura et al.,2013),分支使用邻接法500次bootstrap检验的自引导值,构建系统进化树。文中分析所用网站网址及描述已统计(表2)。
| 图表编号 | XD0031221900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.28 |
| 作者 | 马际凯、成彦丽、仲维平、郝自远、李火根 |
| 绘制单位 | 南京林业大学南方现代林业协同创新中心、南京林业大学南方现代林业协同创新中心、南京林业大学南方现代林业协同创新中心、南京林业大学南方现代林业协同创新中心、南京林业大学南方现代林业协同创新中心 |
| 更多格式 | 高清、无水印(增值服务) |