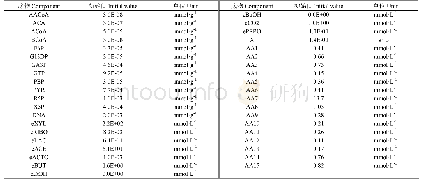
《表1 氨基酸代谢模型参数值》
注:XR:木糖还原酶;rpi A:核糖5-磷酸异构酶A;Pur M:嘌呤代谢;rib A:环化水解酶II;DPO1:二磷酸核苷激酶;TKTa:转酮醇酶;PFK:磷酸果糖激酶;GAPDH:甘油醛-3-磷酸脱氢酶;PGK:磷酸甘油酸激酶;PK:丙酮酸激酶;LDH:乳酸脱氢酶;PFOR:铁氧还蛋白氧化还原酶;PTA:磷
基于Zhao等[10]建立的丙酮丁醇梭菌ABE发酵的碳动态代谢模型,增加氨基酸代谢网络,用以描述玉米浆为底物的氨基酸动态代谢过程。用Michaelis-Menten方程描述了各通量的动力学和细胞的比生长速率。化学反应计量关系、代谢通量动力学方程和质量平衡方程参考Zhao等[10]的研究确定。根据Rizzi等[17]提出的方法确定模型结构和动力学参数,并通过最小二乘法优化模拟结果。通过定义试验值与模拟值之间残差的加权平方和为目标函数,在参数变化范围内优化函数最小值得到参数值。根据在MATLAB中输入模型的参数值(见表1)和代谢物初始值(见表2)进行动态代谢模型模拟得到氨基酸消耗模拟曲线。运用MATLAB R2018b进行动态代谢模型模拟及数据分析,运用Canoco5对数据进行相关性分析。
| 图表编号 | XD00192681500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.01 |
| 作者 | 赵新河、贺壮壮、赵玉斌、马成业、任晓洁 |
| 绘制单位 | 山东理工大学农业工程与食品科学学院、山东理工大学考林腊特列杰微生物脂质国际研究中心、重庆市科学技术研究院、山东理工大学农业工程与食品科学学院、鲁洲生物科技有限公司、山东理工大学农业工程与食品科学学院、山东理工大学农业工程与食品科学学院、山东理工大学考林腊特列杰微生物脂质国际研究中心 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式