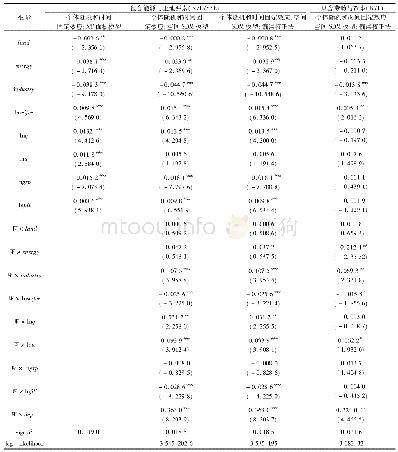
《表4 不同人群个体识别率和累积随机匹配概率统计结果(左侧为CE结果,右侧为NGS结果)》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《15个常染色体STR基因座在中国汉、藏、蒙、维、彝的序列分布差异》
根据各基因座等位基因频率(见附表1),计算个体识别率和累积随机匹配概率。统计结果显示:序列差异引起的等位基因多态性对复杂序列基因座D21S11、D2S1338、D3S1358、D8S1179、vWA,简单序列基因座D13S317、D16S539、D7S820、D5S818个体识别概率均有提高(见表4),平均增长幅度为汉族5.0%(1.9%~8.1%),藏族5.8%(1.7%~13%),蒙古族5.4%(1.5%~11.7%),彝族4.3%(0.4%~6.4%),维族5.2%(2.6%~10%)。基因座个体识别率增长幅度在不同人群表现出一定差异,例如藏族人群在D3S1358和D16S539基因座,蒙古族人群在D21S11基因座,维族人群在D5S818基因座内的DP涨幅明显高于其他人群。
| 图表编号 | XD001512400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.12.20 |
| 作者 | 刘志高、王颖希、李甫、马原、焦章平 |
| 绘制单位 | 内蒙古乌海市公安局刑警支队、北京市公安司法鉴定中心、北京市东城区公安司法鉴定中心、北京市朝阳区公安司法鉴定中心、北京市公安司法鉴定中心 |
| 更多格式 | 高清、无水印(增值服务) |