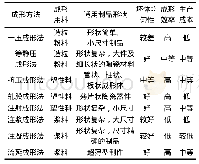
《表2 TADs识别方法:基于生物信息学的Hi-C研究现状与发展趋势》
拓扑相关域TADs是染色体区域内部交互作用水平远高于相邻区域的染色体结构单元,呈嵌套式(domain-in-domain)层级结构(图2C),已被证实广泛存在于真核生物的染色体当中[34,35]。TADs边界富集染色质调控蛋白CTCF、多种组蛋白修饰和持家基因等,其结构内部的基因持有共同的调控元件,如启动子和增强子等,这些基因在多种细胞系中存在协同表达特征,由此形成一个相对独立的调控单元,被认为是复制时间调控(replication-timing regulation)的稳定结构[34,36,37]。因此,TADs是染色体三维结构中的重要高阶结构单元和基因调控单元,对TADs的识别分析有助于理解染色体的复杂结构及其与生物学功能之间的关系。2012年,Dixon等[34]在最先发现接触矩阵中TADs结构的同时,提出一种互作方向指数(directionality index,DI)识别TAD边界点,并首次分析了TADs边界点附近CTCF和组蛋白修饰的高富集度以及基因的高表达水平特征。2014年,Levy-Leduc等[38]提出采用标准动态规划法,迭代求解TADs边界分割模型以得到TADs边界点的HiCseg方法。2015年,上海交通大学Shi联合美国南加州大学Shin等[39],提出TADs边界点识别方法TopDom,其采用钻石形滑动窗口法,提取接触矩阵对角线附近窗口内交互频率的统计特征,将特征曲线的局部极大值作为TADs边界点。2016年,Weinreb等[40]提出基于TADs内部交互频率的经验分布,进行层级式TADs识别的TADtree方法。2017年,Serra等[41]提出采用基于BIC惩罚的最大似然估计求解接触矩阵交互频率的概率模型,识别TADs边界点的TADbit方法。2017年,华中农业大学彭城等[42]提出层级式TADs识别方法HiTAD,其采用基于适应性交互方向指数的隐马尔科夫模型预测TADs边界点,在此基础上,采用迭代最优化策略搜寻接触矩阵中的层级式TADs。2017年,Haddad等[43]提出采用接触矩阵行或列的层次聚类,识别层级式TADs的IC-Finder方法。2017年,Yu等[44]提出采用高斯混合模型,进行层级式TADs识别的GMAP方法。2018年,Norton等[45]提出基于图理论进行层级式TADs识别的3DNetMod方法。2018年,中国科学院北京基因组研究所张治华团队联合北京航空航天大学计算机科学学院软件开发环境国家重点实验室李昂升团队,提出一种基于图结构熵理论的快速层级式TAD识别方法de Doc[46]。2018年,清华大学生物信息学教育部重点实验室陈阳等、南方科技大学前沿与交叉科学研究院李贵鹏等以及美国德克萨斯大学Zhang等[47],提出结合局部相对隔绝指数和多尺度聚类法进行TADs边界点识别的HiCDB方法。各主要TADs识别方法如表2所示。目前,除HiCDB之外,其他方法均不具备不同条件下TADs边界点差异性分析的能力。各方法按照是否考虑TADs的层级式结构又可分为边界点式和层级式两大类,前者如DI、HiCseg、TopDom和TADbit,后者如TADtree、HiTAD、IC-Finder、GMAP和3DNetMod。
| 图表编号 | XD00126841000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.01.01 |
| 作者 | 吕红强、郝乐乐、刘二虎、吴志芳、韩九强、刘源 |
| 绘制单位 | 西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式