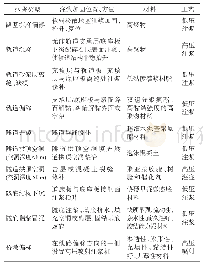
《表3 染色质环识别方法:基于生物信息学的Hi-C研究现状与发展趋势》
染色质环(chromatin loops)也可称为交互峰(interaction peaks),由染色体上相距较远的两个片段构成,其在线性空间中虽相距较远,但在三维空间结构中却具有显著的近距交互作用(图2D)。染色质环对理解染色体结构以及基因表达调控具有重要意义。2009年,Sexton等[48]基于3C技术研究了染色体的空间结构及其在基因表达调控中的作用,在分析染色体显著性交互作用的基础上,提出染色质环概念。2013年,复旦大学遗传工程国家重点实验室田卫东团队将Hi-C染色体空间交互数据引入到人类基因组作用元件与目标基因之间关系的预测当中,结果分析表明,基于Hi-C的染色质环信息能有效提升预测结果的生物功能特性及疾病相关性[49]。随后,染色质环的识别方法不断涌现。2014年,Ay等[50]对Hi-C数据中的随机聚合环和技术型系统偏差进行联合建模分析,提出了染色质环的识别方法Fit-Hi-C。2014年,Rao等[51]基于泊松分布模型提出了HiCCUPS方法,在去除TAD结构影响的前提下预测了染色质交互作用。2014年,Hwang等[52]基于负二项分布概率模型提出一种染色质环识别方法HIPPIE。2015年,Lun等[53]提出包括Hi-C配对末端序列片段预处理,数据标准化以及染色质环识别与差异分析的方法包dffHiC。2017年,中国科学院北京基因组研究所张治华[54]团队针对当时因Kbp分辨率Hi-C数据制备成本高昂而造成染色质环精确识别困难的问题,提出一种结合Kbp分辨率MNaseseq数据和低分辨Hi-C数据的染色质环精确识别方法CISD_loop。2018年,Djekidel等[55]基于空间泊松分布模型提出了检测染色质差异交互作用的方法FIND。随着对染色质环结构的深入了解,国内外相关学者也针对染色质环与病理之间的关系展开研究。2018年,Manduchi等[56]借助功能基因组学数据,分析了二型糖尿病患者基因组中增强子与启动子之间的空间交互及其与基因表达调控之间的关系,证实了增强子–启动子环在该类疾病发生发展中的作用。主要的染色质环识别方法如表3所示。按照针对显著交互作用还是差异交互作用进行鉴别可以划分为两种类型。其中,针对显著交互的有Fit-HiC、HiCCUPS、HIPPIE和CISD_loop,针对差异交互的有DiffiHiC和FIND。
| 图表编号 | XD00126841200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.01.01 |
| 作者 | 吕红强、郝乐乐、刘二虎、吴志芳、韩九强、刘源 |
| 绘制单位 | 西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院、西安交通大学电子与信息工程学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式