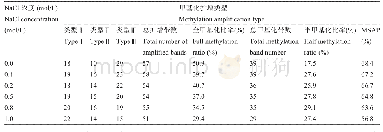
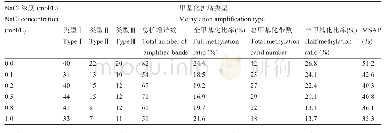
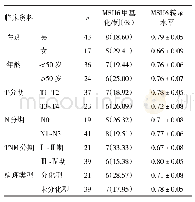
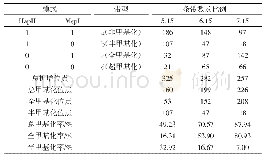
《表2 不同林分锥的MSAP条带类型和甲基化水平》
同行数据后相同字母表示差异不显著(P>0.05)。
为保证选取片段的质量,每对引物在100~500 bp长度内选择25~30个片段,各对引物的位点数分别为E3-H/M2:26、E5-H/M2:34、E6-H/M1:26、E8-H/M1:27、E8-H/M5:29、E9-H/M2:34,共176个片段。用R语言包“msap”分析MSAP矩阵,分析6对引物的扩增片段多态性,依据对Eco RI-Hpa II和Eco RI-Msp I酶切后的表征片段有无的0,1数据表所构成的二进制数据矩阵,进一步统计甲基化敏感位点(MSL)和不敏感位点(NML)的数量、多态率、香浓多样性指数,并对两种位点的香农多样性指数进行Wilcoxon秩检验,比较表观遗传多样性的差异。结合选择性扩增条带的特征,将MSAP矩阵按照DNA甲基化模式进行分类,检测到4种条带类型(表2),其中类型IV不在本研究的范围,只研究未甲基化、全甲基化、半甲基化3种模式[16]。甲基化率的计算公式分别为:半甲基化率(%)=II型/(I型+II型+III型);基化率(%)=III型/(I型+II型+III型);总的甲基化率(%)=(II型+III型)/(I型+II型+III型)。统计3种类型扩增条带的数量,计算锥种群不同模式、不同林分的甲基化水平。利用GenALEX软件进行5种生境的ANOVA分析,并进行显著性检验。使用距离矩阵用Mega 6进行UPGMA聚类分析。所有显著性分析时先检验样本的正态性,当样本都服从正态分布时,运用独立样本t检验,样本不服从正态分布时,运用非参数Mann-Whitney U检验,显著性水平以P<0.05为标准。
| 图表编号 | XD00122345800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.01.01 |
| 作者 | 李文琪、姜楚、欧阳学军、林永标、练琚瑜、刘卫 |
| 绘制单位 | 中国科学院华南植物园、中国科学院大学、中国科学院华南植物园、中国科学院大学、鼎湖山国家级自然保护区管理局、中国科学院华南植物园、广东省应用植物学重点实验室、中国科学院华南植物园、中国科学院华南植物园 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式