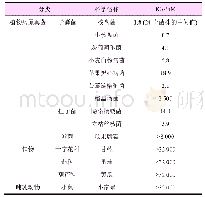
《表1 脚手架链碱基序列:E.coli K-12琥珀酸脱氢酶sdhC基因纳米锥的设计及自组装》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《E.coli K-12琥珀酸脱氢酶sdhC基因纳米锥的设计及自组装》
首先改进DAEDALUS软件中的DX_cage_design模块,使该软件能读取sdh C基因的核酸序列。按照Waston-Crick的B型DNA双螺旋模型,每螺旋为10.5个碱基对时,DNA折叠的应力最小,所以待设计的正四面锥的每条棱应有n10.5bp(n为正整数)个碱基,且由两条订书钉单链和两条脚手架链组成。在DAEDALUS源代码中,加入一个求余函数rem(edge_length,21),检测每条棱的碱基数是否为21bp的整数倍,如果不是,程序则会自动在棱上增加5、10或11个碱基。同理,DAEDALUS输出订书钉链也会尽量接近n21bp,以保证核酸自组装时不产生过度弯曲[11]。理论上DAEDALUS允许输入脚手架链的碱基个数没有限制,但是源代码中的参数均依照M13mp18单链DNA(7249bp)设定。为重新设定计算参数,对sdh C基因序列进行了上下游延伸处理,即在sdh C基因序列上下游各补充117bp,其基因图谱如图1所示,总长为624bp,其中118~507bp为sdh C基因。将该624bp的碱基序列作为脚手架链输入DAEDALUS软件,脚手架链序列如表1所示。经过计算正四面锥各棱含有两组碱基数为52bp的DNA双链,图1中的箭头表示折叠后每条棱对应的碱基位置,AD棱的碱基序列包括(1,33)、(606,624)、(138,189);CD棱包括(34,86)、(503,553);BD棱包括(87,137)、(242,294);AB棱包括(190,241)、(348,399);BC棱包括(295,347)、(452,502);AC棱包括(400,451)、(554,605)。
| 图表编号 | XD00118524100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.02.05 |
| 作者 | 周末、刘彦君、佟欣瑞、董延甲、吴欣瑜、王迎香、应明 |
| 绘制单位 | 天津理工大学化学化工学院、天津理工大学化学化工学院、天津理工大学化学化工学院、天津理工大学计算机科学与工程学院、天津理工大学化学化工学院、天津理工大学化学化工学院、天津理工大学化学化工学院 |
| 更多格式 | 高清、无水印(增值服务) |