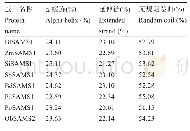
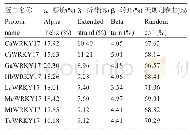
《表3 ClDGAT、ClFAD2和ClSAD蛋白二级结构预测结果(%)》
ClDGAT、ClFAD2和ClSAD蛋白二级结构预测结果表明,3个蛋白的二级结构均含有4种结构类型,且以α-螺旋和无规则卷曲为主(表3)。利用SWISS-MODEL预测3个蛋白的三级结构,结果如图3所示。ClDGAT蛋白的第369~443位氨基酸残基能与D-alanyl转运蛋白DltB模板比对上,其QMEAN4为-1.95(图4-A),GMQE为0.07;ClFAD2蛋白的第89~161位氨基酸残基能与模板酰基-CoA去饱和酶比对上,其QMEAN4为-3.83(图4-B),GMQE为0.08;ClSAD蛋白的第46~393位氨基酸残基能与模板酰基-ACP去饱和酶比对上,其QMEAN4为-1.24(图4-C),GMQE为0.82。3个蛋白三级结构的QMEAN4均大于-4.00,说明采用SWISS-MODEL进行同源建模的质量较好,尤其是ClSAD蛋白三级结构的GMQE接近于1.00,即该结果较可靠。
| 图表编号 | XD00156671100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.03.01 |
| 作者 | 付瑜华、蒙秋伊、李秀诗、杨小雨、敖茂宏、周祥、申刚、刘凡值 |
| 绘制单位 | 贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所、贵州省农业科学院亚热带作物研究所 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式