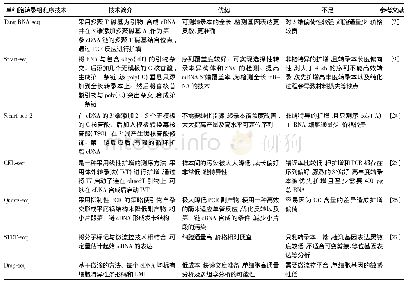
《表1 转录组学测序技术对比》
转录组学不同方法之间的差异主要集中在测序分析上。转录组的测序方法包括两种。一种基于杂交的方法(hybridization-based approaches),其原理是在玻璃,塑料或硅基底制成的芯片上,固定数十万个已知短核苷酸序列,然后与待测样品进行反应,如果待测物中存在互补序列,就会发生杂交。通过荧光标记、表面等离子体共振、比色和拉曼光谱等方法来检测杂交结果[2];另一种是基于测序的方法确定cDNA序列,如基于EST(Expressed Sequence Tag)文库的测序方法。EST是从一个随机选择的cDNA进行5’端和3’端单次测序获得短cDNA的部分序列,用来代表一个完整基因的一部分。基于EST的方法包括基因表达序列分析(serial analysis of gene expression,SAGE)、加帽端分析测序(cap analysis of gene expression,CAGE),以及近些年得到大量应用的RNA-seq测序。RNA-Seq使用下一代测序技术(Nextgeneration sequencing,NGS),由于其先进性得到了迅速发展。几种测序方法的比较见表1。
| 图表编号 | XD00122138300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.09.13 |
| 作者 | 成文龙、张永刚、张亚雷 |
| 绘制单位 | 同济大学环境科学与工程学院(污染控制与资源化研究国家重点实验室) |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式