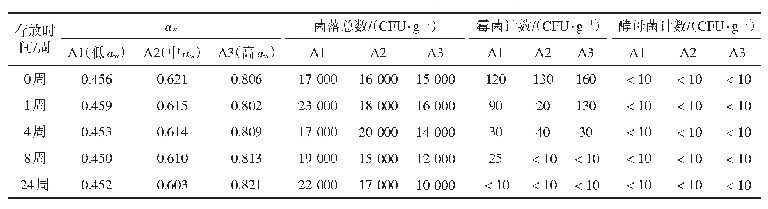
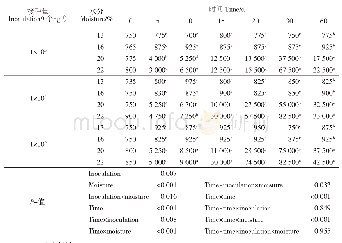
《表2 玉米中霉菌总数PLSR模型预测分析结果》
PLSR建模方法分别以光谱特征波长吸光度、图像颜色特征参数及光谱和图像融合特征参数为自变量,玉米样品菌落总数为因变量,选取2/3样品作为建模集,剩余1/3样品作为验证预测集,其定量分析统计结果如表2所示。Rc2/Rp2值、RPD值越大和RMSEP值越小模型性能较优[31]。由表2可知,基于光谱特征波长吸光度、图像颜色特征参数和光谱及图像融合信息对受单一霉菌侵染的玉米样品进行建模和预测时,Rc2和Rp2分别高于0.920和0.890,RMSEC低于0.522(lg (CFU/g)) ,RMSEP值也较小(0.313~0.728(lg (CFU/g)))。对单一霉菌进行交互验证时,所得RMSECV值也偏低,均小于1.109 (lg (CFU/g))。除基于光谱特征波长的层出镰刀菌195647组外,其余各组模型的RPD值均大于3.0,表明模型具有定量分析应用潜力。对所有霉菌侵染的225份玉米样品和27份菌落总数不为0的对照组样品综合建立PLSR分析模型,共252个样品参与建模。结果表明,3个模型的预测集的Rp2值分别为0.851、0.889和0.894,RMSEP分别为0.823、0.717、0.665 (lg (CFU/g)) ,RPD值分别为2.46、2.93和3.06。从结果可以看出,基于光谱及图像融合特征参数建立的PLSR模型效果普遍优于单一光谱和图像技术建立的模型效果,表明该模型鲁棒性较好,预测精度高,可用于定量分析。图6为基于光谱特征波长吸光度、图像颜色特征参数和光谱特征波长及图像特征参数融合信息的所有252个玉米样品菌落总数实测值与光谱预测值的相关关系图,样品均分布于中心线两侧,表明两者具有较高的相关性。由于样品来源的差异性,模型性能略有下降,但RPD值仍较高,表明模型具有一定的实际检测能力。
| 图表编号 | XD0075204800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.08.25 |
| 作者 | 沈飞、黄怡、周曰春、刘琴、裴斐、李彭、方勇、刘兴泉 |
| 绘制单位 | 南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、南京灵山粮食储备库有限公司、南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、南京财经大学食品科学与工程学院江苏省现代粮食流通与安全协同创新中心、浙江农林大学农业与食品科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式