《表1 柞蚕微孢子基因组LTR反转座子不同家族特征详细统计表》
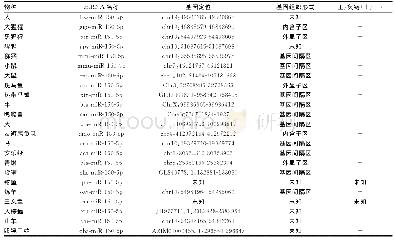
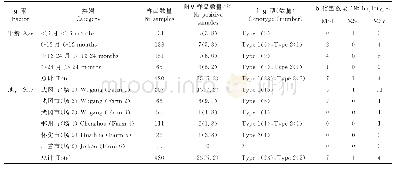
LTR反转座子主要编码Pol多聚蛋白来完成转座功能,该蛋白包含了多个催化结构域,其中以保守性强的RT为最重要特征。根据这一特点并结合反转座子结构的特异性,本研究在柞蚕微孢子虫基因组中鉴定到6个LTR反转座子家族,分别命名为Nar1,Nar2,Nar3,Nar4,Nar5和Nar6。其中,Nar1,Nar2,Nar3,Nar4和Nar5家族含有完整的转座子家族所具有的特征性序列即反向末端重复(Inverted terminal repeat)和靶位点重复序列(表1)。研究结果还显示:柞蚕微孢子虫LTR反转座子的长度在3~4kb之间;大部分家族的LTR末端核苷酸具有一致性,为TGT/ACA或TG/CA;但家族间的靶位点重复序列在核苷酸数量和种类上则具有差异,长度为5~6个核苷酸(表1)。此外,通过RepeatMasker软件对LTR转座子在柞蚕微孢子虫基因组上的拷贝个数进行了初步的统计,发现不同家族间拷贝子数量相差较大,其中拷贝子数量最多的为Nar3和Nar4家族,均为16个;而Nar2家族拷贝子数量最少,只有3个(表1)。
| 图表编号 | XD0053709700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.05.25 |
| 作者 | 周俊、石鹏、张爽、许金山、张小燕 |
| 绘制单位 | 重庆师范大学重庆市媒介昆虫重点实验室重庆市动物生物学重点实验室、重庆师范大学重庆市媒介昆虫重点实验室重庆市动物生物学重点实验室、重庆师范大学重庆市媒介昆虫重点实验室重庆市动物生物学重点实验室、重庆师范大学重庆市媒介昆虫重点实验室重庆市动物生物学重点实验室、重庆师范大学重庆市媒介昆虫重点实验室重庆市动物生物学重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式