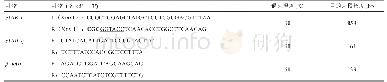
《表1 参考物种信息:绵羊角蛋白关联蛋白2基因的克隆表达与生物信息学分析》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《绵羊角蛋白关联蛋白2基因的克隆表达与生物信息学分析》
扩增产物经1.0%琼脂糖凝胶电泳检测,用胶回收试剂盒回收目的片段,取PCR回收产物4μL,pMD19-T载体1μL与5μL的SolutionⅠ混匀,4℃过夜,再将质粒pMD19-T-KAP2转化大肠杆菌DH5α感受态细胞,涂布于氨苄抗性的固体LB平板,37℃培养12~16h。经蓝白斑筛选后,接种到氨苄抗性的LB液体培养基中,37℃继续培养,取菌液用DNA质粒提取试剂盒提取质粒,操作步骤按试剂盒进行,对重组质粒进行BamHⅠ和SalⅠ双酶切及PCR鉴定。PCR酶切体系20μL:pMD19-T-KAP2重组质粒8μL,BamHⅠ、SalⅠ各1μL,10×Buffer 2.0μL,ddH2O 8μL,预计扩增片段大小为463bp。阳性产物送宝生物工程(大连)有限公司进行测序,并对测序结果进行生物信息学分析。1.2.6生物信息学分析采用EditSeq(DNAS-tar)对绵羊KAP2基因进行碱基组成分析;参考GenBank中的7种不同物种KAP2基因序列,采用MegAlign(DNAStar)进行各物种KAP2基因的同源性分析,并构建系统进化树,参考物种信息见表1;采用ProtParam(http:∥web.expasy.org/protparam/)对绵羊KAP2基因编码蛋白基本理化性质进行分析,并采用TMpred(http:∥www.ch.embnet.org/software)对KAP2蛋白跨膜区及跨膜方向进行预测;用在线GOR4工具对KAP2蛋白的二级结构进行预测,并用Protean(DNAStar)中的最优方法对克隆序列编码蛋白的二级结构进行分析,进一步利用SWISS-MODEL(https:∥swissmodel.expasy org/)预测三级结构。
| 图表编号 | XD0022137200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2018.06.20 |
| 作者 | 刘晓军、张立春、韩林凯、楚熹橦、崔成都、鲁承、金海国、孙福亮 |
| 绘制单位 | 延边大学农学院、吉林省农业科学院畜牧分院、延边大学农学院、延边大学农学院、延边大学农学院、延边大学农学院、吉林省农业科学院畜牧分院、延边大学农学院 |
| 更多格式 | 高清、无水印(增值服务) |