《表1 不同噬菌体预测工具及其部分特征》
“–”:代表无。
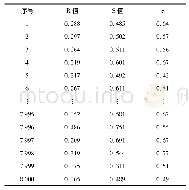
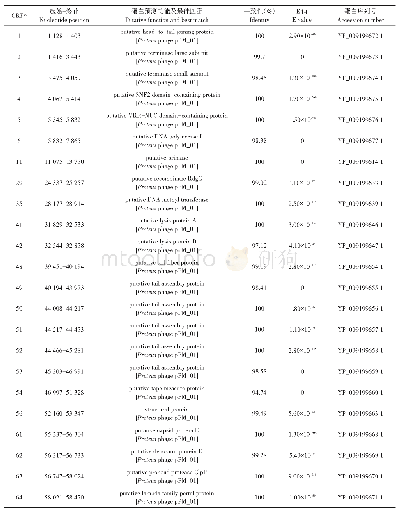
随着计算机生物信息学技术的广泛运用、新测图1噬菌体的不同生命循环周期序细菌基因组数量的不断增加以及人们对前噬菌体序列在细菌DNA中所占比例的认识,研究者开发出了预测细菌基因组中前噬菌体的软件,见表1。早期开发的Phi Spy软件通过监测与测序基因组没有相似性的噬菌体特征来识别噬菌体[6],Vir Sorter利用已知病毒数据库中的比对和相似性搜索预测宏基因组数据中病毒的重叠群[7];Vir Finder用于从宏基因组学数据中识别原核病毒序列,其基于k-mer特征的机器学习来预测重叠群是否为噬菌体[8];Meta Phinder通过与全基因组噬菌体序列数据库进行比较,在宏基因组学数据库中识别噬菌体起源的组装基因组片段(即重叠群)[9];PHASTER用于快速识别和注释细菌基因组和质粒中的噬菌体序列[10];2018年,Amgarten等[11]开发了一种预测宏基因组库中双链DNA噬菌体序列的工具MARVEL,并将MARVEL的性能与Vir Sorter和Vir Finder进行比较,发现尽管3个程序都有很好的特异性,但MARVEL灵敏度更高,能够在宏基因组文库中鉴定出比以前更多的噬菌体序列;近年来开发了Prophage Hunter,它提供一站式web服务,从细菌基因组中提取前噬菌体基因组,评估前噬菌体的活性,识别系统发育相关的噬菌体,并注释噬菌体蛋白质的功能[12]。
| 图表编号 | XD00213926800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2021.03.01 |
| 作者 | 陈学梅、魏云林、季秀玲 |
| 绘制单位 | 昆明理工大学生命科学与技术学院、昆明理工大学生命科学与技术学院、昆明理工大学生命科学与技术学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式