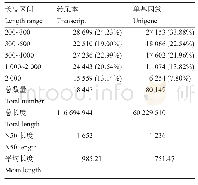
《表1 转录本及基因序列拼接长度分布》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《黄麻(Corchorus capsularis L.)干旱胁迫转录组初步分析》
本次转录组测序经去除接头以及低质量序列信息等后,共获得7.08 G高质量测序数据(Clean reads),并使用Trinity软件(Grabherr et al.,2011)对这些Clean reads进行拼接共得到45 942条转录本(Transcripts)。随后,使用Corset软件(Davidson and Oshlack,2014),利用比对上转录本的reads数和表达模式对转录本进行层次聚类,从而得到23 297个非冗余的基因(Unigenes)。对这些转录本和基因序列长度进行统计发现,二者具有相似的序列长度分布规律(图1A;图1B),且不同长度范围内的基因数目均少于转录本数目(图1C)。然而,基因和转录本在序列长度分布统计方面没有明显差别(表1),说明从转录本至基因的序列冗余是均匀分布在不同长度序列上。
| 图表编号 | XD00189158400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.12.14 |
| 作者 | 金关荣、陈杰、安霞 |
| 绘制单位 | 浙江省萧山棉麻研究所浙江省农业科学院花卉研究开发中心、华中农业大学生命科学技术学院、浙江省萧山棉麻研究所浙江省农业科学院花卉研究开发中心 |
| 更多格式 | 高清、无水印(增值服务) |