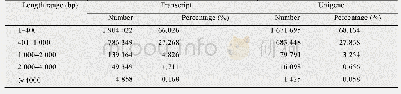
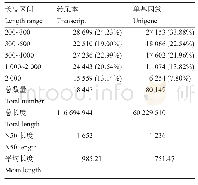
《表1 拼接转录本和基因序列长度频数分布》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《牡丹(Paeonia suffruticosa Andr.)根组织差异表达基因分析》
通过Illumina HiSeqTM4000高通量测序平台对试验区6年生凤丹(Paeonia ostii'Phoenix White')和冰山雪莲(Paeonia rockii'Bing Shan Xue Lian')2个品种幼根组织进行转录组测序,得到原始序列数据raw reads,然后对raw reads进行过滤,去掉带有接头、低质量和无法确定碱基信息的reads,得到测序序列数据Clean reads,其中表征数据质量的Q20和Q30大于90%,GC含量在45%左右,数据质量达到后续分析要求;同时采用Trinity软件对不同长度的转录本clean reads进行拼接处理,得到转录本拼接文件Trinity fasta,从同一个基因的不同转录本中挑选出最长的转录本作为该基因的Unigene,最后得到276043条Unigene,以此进行后续注释分析。转录本和Unigene长度分布频数显示(表1),转录本和Unigenes的长度主要分布在200~1 000 bp之间,转录本transcript平均长度583 bp,Unigene平均长度837 bp,反映拼接效果的N50长度分别为798 bp和1 064 bp,大于其平均长度,表明拼接片段的完整性较好。
| 图表编号 | XD0058974300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.05.28 |
| 作者 | 李锦馨、毕江涛、马燕 |
| 绘制单位 | 宁夏大学农学院、宁夏大学环境工程研究院、宁夏大学农学院 |
| 更多格式 | 高清、无水印(增值服务) |