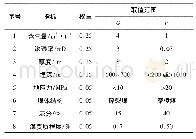
《表4 10种蛋白质结构在Ramachandran图中的有利区域和不利区域的百分比(%)》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《压缩感知理论(CS)在弱信号量子测量与蛋白质结构计算中的应用》
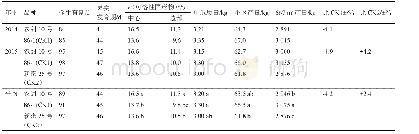
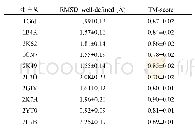
选取10个不同大小、不同拓扑结构的蛋白进行重建,结果如表3和图27所示[54].表3显示与PDB中的蛋白质结构比较,利用矩阵填充重建的蛋白质结构的RMSD在2?左右,说明该重建总体上是准确的,另外一个评价蛋白质结构相似性的指标TM-score大部分在0.8以上,同样也说明重建方法的可行性.图27则展示了相应的蛋白结构,可以看到,计算结构和参考结构之间有很高的重合.通过进一步分析了重建蛋白的主链原子在Ramachandran图上的分布(表4,图28(a)和(b)),可以发现重建蛋白的原子大部分都处在Ramachandran图的有利区域,说明了重建蛋白结构的合理性.最后通过分析矩阵填充方法重建的蛋白质的二级结构,发现与PDB的参考结构具有高度的相似性(如图28(c)所示),反映了计算结构的稳定性.至此,通过一个小的测试集,说明了MC算法可用于真实NMR测量的蛋白质结构重建,且重建效果与PDB的传统重建方法相当.
| 图表编号 | XD00149587100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.01 |
| 作者 | 彭绪彪、赵清、葛墨林 |
| 绘制单位 | 北京理工大学物理学院量子技术研究中心、北京理工大学物理学院量子技术研究中心、北京理工大学物理学院量子技术研究中心、南开大学陈省身数学所 |
| 更多格式 | 高清、无水印(增值服务) |