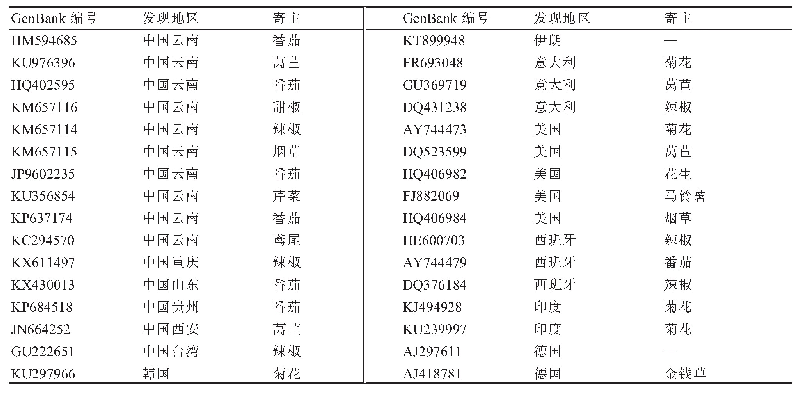
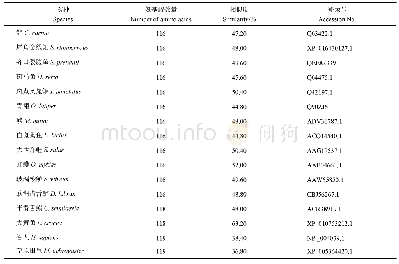
《表2 LH氨基酸序列比对及系统发育进化树构建的参照物种信息》
将获得的正向和反向序列在NCBI数据库中进行同源性比对,使用SeqMan Pro Version 7.1.0对中间片段序列、正向和反向序列进行剪切拼接(周惠强,2019),以获得大刺鳅LH基因全长cDNA序列。使用NCBI数据库中的BLAST对大刺鳅LH基因全长cDNA序列进行同源性比对,以DNAMAN 6.0推导其氨基酸序列;从GenBank中选择硬骨鱼类、两栖动物、鸟类和哺乳动物等物种的LH氨基酸序列(表2),应用ClustalX 2.1进行LH氨基酸序列比对,具体操作为:选择比对模型Multiple Alignment Mode(多序列比对),默认参数设置,选用Do Complete Alignment(进行完全比对),比对结果以BOXSHADE Server进行着色处理。采用Multiple Sequence Alignment by CLUSTALW进行物种同源性比对,并以MEGA 6.0中的邻接法(Neighbor-joining,NJ)构建系统发育进化树,选择模型为Poisson Correction(泊松修正),自引导检验(Bootstrap)设为1000。
| 图表编号 | XD00147649700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.07.30 |
| 作者 | 黄小琪、钟东明、何佩莹、张明清、周惠强、舒琥 |
| 绘制单位 | 广州大学生命科学学院、广州大学生命科学学院、广州大学生命科学学院、广州大学生命科学学院、广州大学生命科学学院、广州大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式