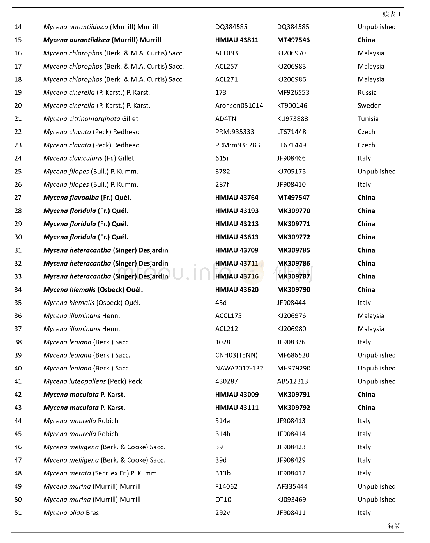
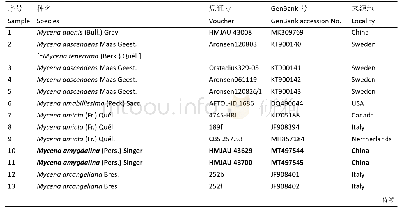
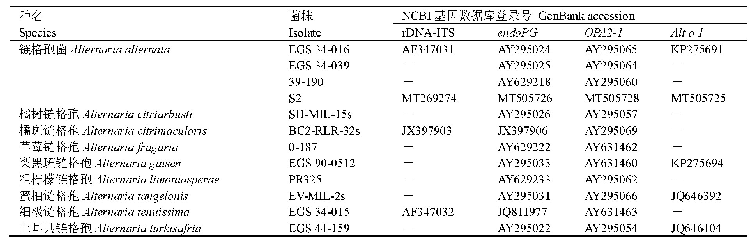
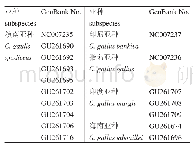
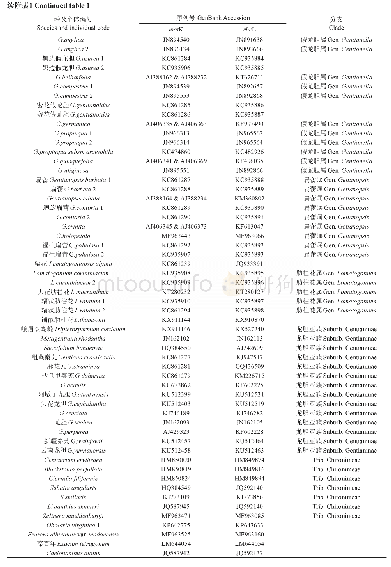
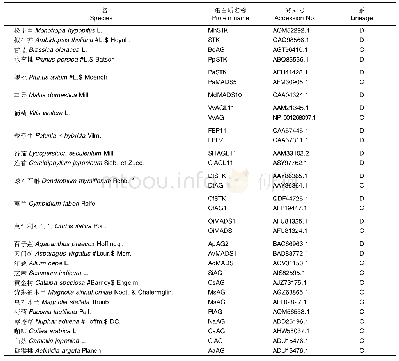
《表2 用于序列比对和系统发育分析的物种信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《甜荞FaesSTK基因在长花柱长雄蕊突变体lpls中的表达分析》
将甜荞FaesSTK同源基因的开放阅读框(open reading frame,ORF)编码的氨基酸序列在NCBI数据库(https://blast.ncbi.nlm.nih.gov/Blast.cgi)中进行BlastP同源比对搜索,选取20种来自不同植物的STK同源蛋白(表2),采用MEGA 5.0软件的邻接法(Neighbor-Joining,NJ)构建蛋白序列的分子系统发育树,同时选取STK(拟南芥,OAO98566.1)、PpSTK(水蜜桃Prunus persica(L.)Batsch,ABQ85556.1)、FPB11(矮牵牛Petunia×hybrida Vilm.,CAA57445.1)、TAGL11(番茄,AAM33102.2)等7个物种的8个同源蛋白,采用Bioedit 7.0软件中的Clustal W程序,对3种花型的甜荞FaesSTK转录因子的结构域进行分析。
| 图表编号 | XD00156778500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.08.01 |
| 作者 | 李逸、王璇、刘志雄 |
| 绘制单位 | 长江大学园艺园林学院、长江大学园艺园林学院、长江大学园艺园林学院 |
| 更多格式 | 高清、无水印(增值服务) |