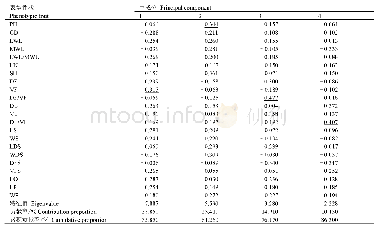
《表2 草果不同居群遗传多样性的SRAP分析》
注:JP:金平居群;PB:屏边居群;YY:元阳居群;LVC:绿春居群;PPB:多态性条带比率;Na:观测等位基因数;Ne:有效等位基因数;H:Nei's基因多样度;I:Shannon信息指数;Ht:总遗传多样性;Hs:居群内遗传多样性;Gst:遗传分化系数
利用POPGENE 1.32软件对云南4个草果居群进行了遗传多样性分析。在物种水平上,云南草果的观测等位基因数(Na)为1.995,有效等位基因数(Ne)为1.377,多态性条带比率(PPB)为99.45%,Nei's基因多样性指数(H)为0.234,Shannon信息指数(I)为0.369(表2)。在群体水平上,不同草果居群的多态性条带百分率(PPB)从72.93%到77.35%,平均为75.55%;每个位点的观测等位基因数(Na)从1.729到1.774,平均为1.756;每个位点的有效等位基因数(Ne)从1.311到1.399,平均为1.354;Nei's基因多样性指数(H)从0.197到0.234,平均为0.216;Shannon信息指数(I)从0.312到0.357,平均值为0.334。其中金平县草果居群的PPB、Na、Ne、H和I最高,元阳居群的PPB和Na最低,绿春居群的Ne、H和I最低。
| 图表编号 | XD00146670300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.08.28 |
| 作者 | 马孟莉、张婷婷、朱玉逍、齐国芳、雷程亮、王田涛、卢丙越 |
| 绘制单位 | 红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点实验室、红河学院生命科学与技术学院云南省高校滇南特色生物资源研究与利用重点 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式