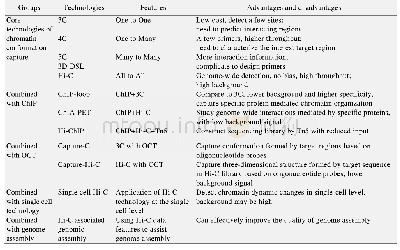
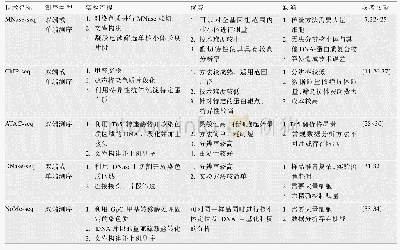
《表1 5种染色质可及性研究技术介绍》
染色质开放区域的研究源于人们发现某些染色质特定位点表现出对DNase I酶切的高度敏感性[12~15]。后期研究表明,这些DNase I敏感位点(deoxyribonuclease I hypersensitive site,DHS)通常是顺式调控元件所在区域[16],其染色质裸露、结构疏松,可与转录因子结合,从而便于DNase I与之结合并剪切,进而表现出高度敏感性[17]。基于上述原理,染色质开放区域的鉴定工作也随之展开。最先开展的是DHS鉴定分析工作,该分析依赖DNase I高度敏感性特点,并与Southern杂交技术结合,不过很快发现该方法的灵敏性和精确性都较低,并且耗时费力[18,19]。随着高通量测序技术(high-throughput sequencing,HTS)的发展及测序成本不断降低,衍生出一系列研究染色质开放区域的技术与方法,如脱氧核糖核酸酶I超敏感位点测序(deoxyribonuclease I hypersensitive site sequencing,DNase-seq)[20]、微球菌核酸酶敏感位点测序(micrococcal nuclease sequencing,MNase-seq)[11]、甲醛辅助性调控元件分离测序(formaldehyde-assisted isolation of regulatory elements followed by sequencing,FAIRE-seq)[21]、核小体定位和甲基化组测序(nucleosome occupancy and methylome sequencing,NOMe-seq)[22]和ATAC-seq。在上述5种技术中,获取染色质开放信息的方式分为3种:DNase-seq、MNase-seq以及ATAC-seq采用酶切法;FAIRE-seq采用物理断裂法;NOMe-seq技术则利用甲基化修饰。5种技术的具体信息见表1。ATAC-seq与其他4种技术相比表现出更为简便和高效的优势,一经发明就被广泛采用,成为当前染色质开放区域获取的前沿技术。下面将对上述染色质开放区域获取技术的发展历程、作用机理以及研究进程进行描述。
| 图表编号 | XD00140605500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.04.01 |
| 作者 | 吴杰、全建平、叶勇、吴珍芳、杨杰、杨明、郑恩琴 |
| 绘制单位 | 华南农业大学动物科学学院国家生猪种业工程技术研究中心、华南农业大学动物科学学院国家生猪种业工程技术研究中心、华南农业大学动物科学学院国家生猪种业工程技术研究中心、华南农业大学动物科学学院国家生猪种业工程技术研究中心、华南农业大学动物科学学院国家生猪种业工程技术研究中心、仲恺农业工程学院动物科技学院、华南农业大学动物科学学院国家生猪种业工程技术研究中心 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式