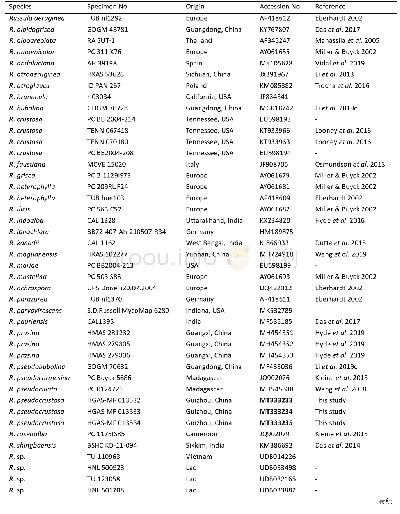
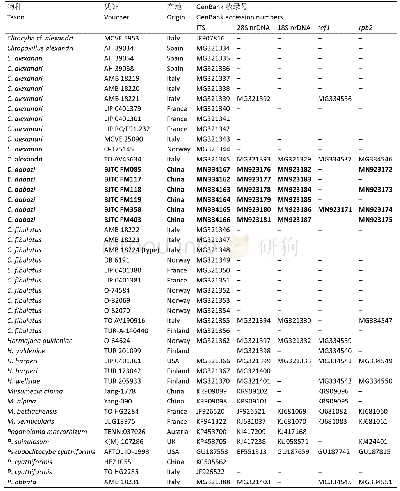
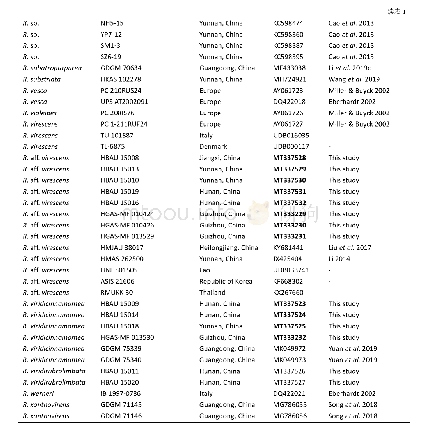
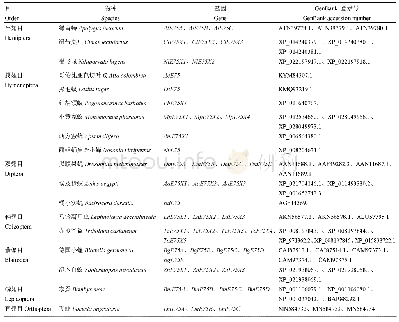
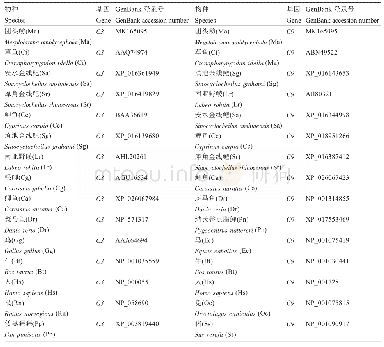
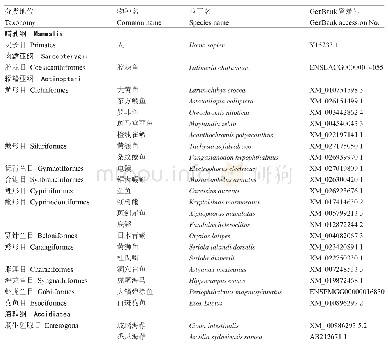
《表3 本研究使用的GP序列的物种分类地位及登录号》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《大弹涂鱼肝型GP基因的克隆及其在氨氮胁迫下的响应》
通过NCBI BLAST搜索程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)和DNAMAN 6.0分析新获得的序列的核苷酸和推导的氨基酸序列(韩雪晴等,2019)。通过EXPASY(https://web.expasy.org/cgibin/compute_pi/)预测蛋白质分子量和等电点(马芳等,2019)。结构域利用SMART网站来进行分析(http://smart.embl-heidelberg.de/)。使用GENEDOC进行多序列比对,通过Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行同源性比较。本研究用于GP进化研究的物种和登录号见表3。
| 图表编号 | XD00136865100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.03.25 |
| 作者 | 郭婷婷、孟繁星、黎明、王日昕 |
| 绘制单位 | 宁波大学海洋学院、宁波大学海洋学院、宁波大学海洋学院、宁波大学海洋学院 |
| 更多格式 | 高清、无水印(增值服务) |