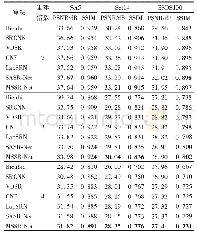
《表4 不同方法在Gram-positive数据集上的比较结果》
%
从表4中可以看到,对于Gram-positive数据集,多特征结合方法取得的总体准确率达到了97.8%,比氨基酸组成方法和自相关系数方法分别高出21.5%和3.0%。需要指出的是,对plasma membrane位点,自相关系数方法和多特征结合方法的敏感性都达到了100%,而氨基酸组成方法仅为82.8%,这说明与简单的氨基酸组成信息相比,氨基酸的位置信息和序列内部不同距离氨基酸间的相互作用信息对蛋白质亚细胞定位预测的准确率影响更大。而本文的多特征结合方法全面优于其他两种方法也说明构建包含更多信息的蛋白质表达模型对提高预测准确率有很大帮助。
| 图表编号 | XD0067427300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.06.01 |
| 作者 | 刘清华、赖裕平、丁洪伟、杨志军、崔晓龙 |
| 绘制单位 | 云南大学信息学院、北方工业大学计算机学院、云南大学信息学院、云南大学信息学院、云南大学微生物研究所 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式