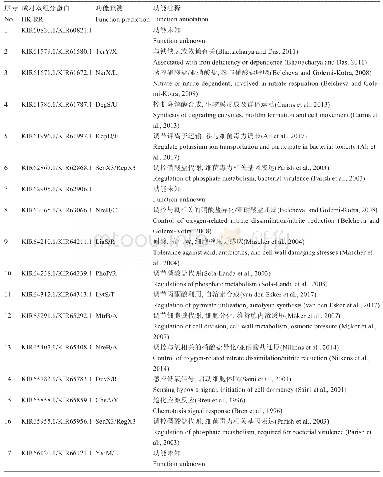
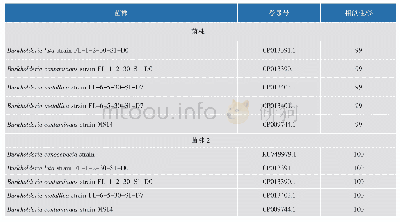
《表2 PDB中预测的各亚基同源性比较》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《嗜碱盐单胞菌X3中异化型硝酸盐还原酶编码基因簇的功能验证及蛋白结构预测》
结合图6和表2分析NarGYJV的蛋白3D结构预测,Phyre2预测的NarG、NarY和NarV蛋白3D结构(见图6 (a)、(b)、(c)) ,在PDB中同源关系最近的是1Q16,来自大肠杆菌三亚基与1Q16之间均具有6%~100%的覆盖率,54%~74%的一致性,各对应区域如图6(d)所示;图6(e)是NarJ的蛋白3D结构预测图,在PDB中预测的唯一同源性3D结构来自家牛(Bos Taurus)的蛋白细胞色素B5(Cytochrome B5)(见图6(f)) ,但其覆盖率仅13%,相似性仅44%(见表2),可能因为PDB库中相应蛋白质数据不全导致比对结果不理想。
| 图表编号 | XD0039106300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.01 |
| 作者 | 王越、李秋芬、张艳 |
| 绘制单位 | 上海海洋大学水产与生命学院、农业部海洋渔业可持续发展重点实验室中国水产科学研究院黄海水产研究所、农业部海洋渔业可持续发展重点实验室中国水产科学研究院黄海水产研究所 |
| 更多格式 | 高清、无水印(增值服务) |