《Table 6 RMSDs of geometric structure between force fields and M06-2X/6-311++G**method》
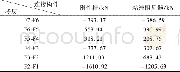
在经OPLS/AA和AMBER OL15优化后的模型分子结构中,可发现有部分氢键复合物中氨基酸的位置发生了明显改变,具体变化情况示于图6,原有氢键被破坏,氨基酸侧链与碱基作用位置改变,形成T型结构,甚至AMBER OL15力场下G1Cys已不属于氢键复合物,因此FFF无法完全模拟体系间氢键作用.无论是氢键键长还是氢键键角,ABEEMσπPFF均得到了与M06-2X/6-311++G**较一致的结果.供体原子X与受体原子Y间的氢键键长和供体原子H与受体原子Y间的氢键键长的RMSD均为0.0094 nm,约占QM下各自类型氢键平均氢键键长(0.29和0.20nm)的3.2%和4.7%;氢键键角的RMSD为4.8°,约占QM下平均氢键键角(152.2°)的3.1%,所有氢键结构偏差均在允许范围内.ABEEMσπPFF下具体每个结构的氢键键长和氢键键角与QM的RMSD值列于表S3(见本文支持信息).
| 图表编号 | XD0031440700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.02.10 |
| 作者 | 许炎、刘翠、韩成娟、潘明玉、孙照琦、韩冰玉、杨忠志 |
| 绘制单位 | 辽宁师范大学化学化工学院、辽宁师范大学化学化工学院、辽宁师范大学化学化工学院、辽宁师范大学化学化工学院、辽宁师范大学化学化工学院、辽宁师范大学化学化工学院、辽宁师范大学化学化工学院 |
| 更多格式 | 高清、无水印(增值服务) |
查看“Table 6 RMSDs of geometric structure between force fields and M06-2X/6-311++G**method”的人还看了
-
- Table 2–Weight, thickness, contact angle, polar force, dispersive force and surface free energy of PP-films and PN-films
-

- Table 6 Path analysis between amount of green manure returning to field and medium tobacco proportion
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式