《表1 ABE碱基编辑系统优化表》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于CRISPR/Cas系统的单碱基编辑技术研究进展》
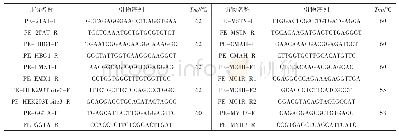
如今已经开发了几种版本的ABE,从ABE1.2发展至ABE7.10[6]。第一代ABE碱基编辑器是利用能直接作用于DNA的突变体Tad A*(D108N和A106V)构建而成的,命名为ABE1.2,但在哺乳动物细胞中的碱基编辑效率非常低,仅为3.2%;在ABE1.2基础上继续通过随机文库筛选新的突变位点,将Tad A上的2个点突变D147Y和E155V引入ABE1.2,构建出ABE2.1系统,碱基编辑效率提高到11%。接下来,在ABE2.1系统中融合表达wt Tad A或Tad A (2.1)*,分别构建出ABE2.9和ABE2.10,由于Tad A以同源二聚体的形式发挥作用,其中一个Tad A催化脱氨,而另一个Tad A作为催化底物tRNA有关位点,ABE2.9能将编辑效率有效提升至24%左右;在此基础上进一步引入突变,将Tad A上的3个新的点突变位点L84F、H123Y和I157F引入ABE2.9得到ABE3.1,效率可提高至29%±2.6%;将Tad A上的4个点突变H36L、R51L、S146C和K157N引入ABE3.1得到ABE5.1,通过大肠杆菌抗性筛选体系进一步优化了Tad A序列,虽能在细菌产生高效定点突变,但是在哺乳动物细胞中的突变效率却降低了;将ABE5.1两个Tad A亚基中N端无催化功能的亚基替换为wt Tad A,形成ABE5.3编辑系统,将编辑效率提升至22%~33%;ABE6.3通过已有突变的重新组合,以去除不利编辑效率达47%±5.8%(表1);ABE7.10编辑系统继续引入突变以解决上述问题,其编辑效率显著提高,在人类细胞中编辑效率约为50%,还可保证极高的精确度和较低的脱靶效应[6](图11)。
| 图表编号 | XD00208040000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.12.01 |
| 作者 | 王玥、牟彦双、刘忠华 |
| 绘制单位 | 东北农业大学黑龙江省动物细胞与遗传工程重点实验室、东北农业大学黑龙江省动物细胞与遗传工程重点实验室、东北农业大学黑龙江省动物细胞与遗传工程重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |