《表7 Plant proteins数据集上四种方法的实验性能对比》
单位:%
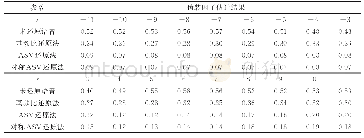
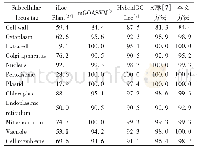
为了进一步验证本文方法的优越性,继续在数据集Plant proteins上进行实验分析,将各位点标签上得到的预测结果与其他现有算法模型取得的实验结果进行比较,其对比结果如表6所示。由表6可知,与传统的蛋白质定位算法i Loc-Plant相比,本文方法在蛋白质各位点标签上的预测准确率有显著的提升。而相较于m GOASVM和Hybrid GO-Loc这两种算法,本文方法除了在Cell wall proteins和Mitochondrion proteins位点的预测准确率稍有下降以外,其他位点的预测准确率均有一定程度的提高;与文献[7]算法相比,本文方法除了在Nucleus proteins位点预测准确率稍有下降,其他位点的预测准确率都基本提升和持平。特别的,本文方法在Extracell proteins、Peroxisome proteins、Plastid proteins和Vacuole proteins位点上取得了100%的预测准确率。由于这四种方法各位点亚细胞预测准确率较为接近,为了进一步验证本文方法的有效性,接下来引入多标签预测评估指标,对四种方法进一步分析比较,其详细对比结果如表7所示。
| 图表编号 | XD00189111600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.11.10 |
| 作者 | 王艺皓、丁洪伟、李波、保利勇、张颖婕 |
| 绘制单位 | 云南大学信息学院、云南大学信息学院、云南大学信息学院、云南大学信息学院、云南大学信息学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式