《表5 Viral proteins数据集上三种方法的实验性能对比》
单位:%
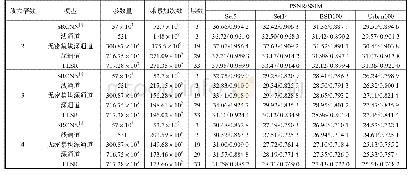
由表4可以看出,本文所提新方法与i Loc-Virus、KNN-SVM、m GOASVM算法的实验结果相比,均有较明显的提高。特别是与文献[7]算法相比,本文方法在Host cell membrane、Host cytoplasm、Host nucleus和Host endoplasmic reticulum位点上的预测准确率均有不同程度的提升,说明了本文在特征融合后引入SDAE深度网络的有效性和科学性。为了进一步验证新方法的优越性,将预测结果与现有算法中表现较好的m GOASVM和文献[7]算法进一步进行比较,其详细对比结果如表5所示。
| 图表编号 | XD00189111300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.11.10 |
| 作者 | 王艺皓、丁洪伟、李波、保利勇、张颖婕 |
| 绘制单位 | 云南大学信息学院、云南大学信息学院、云南大学信息学院、云南大学信息学院、云南大学信息学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式