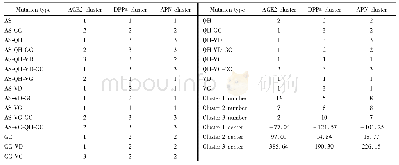
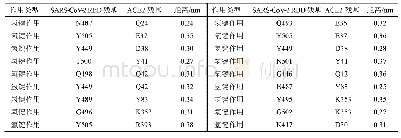
《表1 SARS-CoV-2 S蛋白和ACE2的相互作用方式》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
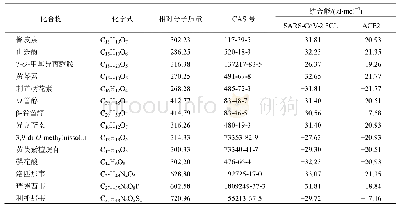
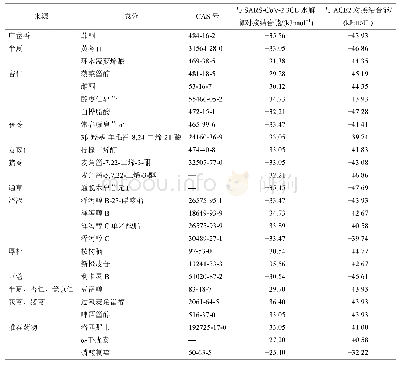
《基于分子对接技术筛选新型冠状病毒S蛋白-血管紧张素转化酶2小分子抑制剂》
下载周强课题组发表的RBD-ACE2-B0AT1复合体晶体结构(PDB:6M17),删除B0AT1部分,去除水分子,利用Macromolecules/Prepare Protein/CleanProtein对蛋白分子进行预处理。利用DS2019中Calculate Mutation Energy(Binding)功能对ACE2蛋白-病毒S蛋白结合面涉及到的氨基酸残基(表1)进行基于相互作用力的虚拟氨基酸突变,如果突变能在-2.09~2.09 kJ/mol,那么Effect为neutral,即这种突变对于亲和力没有影响;如果突变能在2.09 kJ/mol以上,那么Effect为destabilizing,即这种突变会导致亲和力降低,相互作用关系减弱;如果突变能-2.09 kJ/mol以下,那么Effect为stabilizing,即这种突变会导致亲和力上升,相互作用关系增强。将mutation energy大于2.09 kJ/mol的氨基酸残基确定活性位点中的关键氨基酸残基。选择关键氨基酸残基后,利用程序中Receptor-Ligand Interactions模块Define and EditBinding Site/From Current Selection,确定结合空腔(SBD_Site_Sphere)。
| 图表编号 | XD00178403600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.05.12 |
| 作者 | 宋磊、毕思伟、古君、吴童怡、何黎黎 |
| 绘制单位 | 西南民族大学药学院、四川大学华西医院烧伤整形外科、四川大学华西医院心脏大血管外科、西南民族大学药学院、西南民族大学药学院 |
| 更多格式 | 高清、无水印(增值服务) |