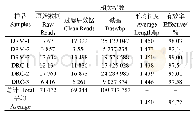
《表1 测序克隆中插入的外源DNA位置信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《利用高效的大片段DNA回收方法改良Fosmid文库的构建》
由于28-45 kb的外源DNA片段包装后可高效进入λ噬菌体头部,本研究将第1至3孔的DNA混合,按照Williams等[30]的方法成功地构建了Fosmid文库。为了初步检测文库质量,随机挑选文库的10个单克隆,提取少量质粒DNA,用限制性内切酶Aat II和Eco72I作双酶切后进行PFGE分析发现,10个质粒均有外源DNA插入,文库阳性率为100%。但是,质粒的酶切产物条带过多,难以推测准确的外源DNA长度(数据未展示)。为了检测文库中外源DNA片段的准确大小,我们又随机挑选了25个单克隆进行双末端Sanger测序并成功获得了24个克隆的双末端序列,这些序列与蜀恢498的基因组序列[18]比对结果如表1所示,插入的外源DNA片段大小在23 kb-52 kb之间,每个克隆的平均插入片段大小约为37.9 kb,标准差为5.2 kb,片段大小比较集中。
| 图表编号 | XD00171460800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.07.26 |
| 作者 | 张翠翠、赵胜、王越、张鹏、常玉晓 |
| 绘制单位 | 广西大学生命科学与技术学院、中国农业科学院深圳农业基因组研究所、广西大学生命科学与技术学院、中国农业科学院深圳农业基因组研究所、广西大学生命科学与技术学院、中国农业科学院深圳农业基因组研究所、中国农业科学院深圳农业基因组研究所 |
| 更多格式 | 高清、无水印(增值服务) |