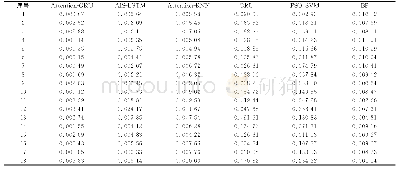
《表2 各脑组织前200个基因表达量预测模型平均绝对误差》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于全血基因表达谱的脑组织基因表达量预测模型的建立》
各脑组织与全血配对可用的样本量最小只有50。为确定特征选择方案,将前200个基因预测任务作为预实验。每个脑组织的前200个基因表达量预测任务分别提取5、10、15和20个最相关的全血基因表达量特征,基于全血基因表达量的LSR脑组织基因表达量预测模型的MAE结果汇总详见表2。对于杏仁核(amygdala)和黑质(substantia nigra)两个样本量最小(n=50,表1)脑组织,提取25个最相关特征的预测模型达到最优预测性能,提取少于25个特征处在欠拟合区域,提取多于25个特征则处在过拟合区域;其他10个脑组织在30个最相关特征时达到最优性能(表2中以*表示),仍处于欠拟合区域。为保证脑组织最小样本量是特征数的4倍左右,统一选择提取15个最相关特征的方案(表2中以Δ表示)。重新用全部样本训练线性回归模型,此线性模型得到的脑组织前200个基因表达量的预测值与真实值的拟合度良好(图1)。
| 图表编号 | XD0093231800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.10.21 |
| 作者 | 徐文剑、李巍 |
| 绘制单位 | 国家儿童医学中心首都医科大学附属北京儿童医院遗传与出生缺陷防治中心北京市儿科研究所出生缺陷遗传学研究北京市重点实验室儿科重大疾病研究教育部重点实验室、国家儿童医学中心首都医科大学附属北京儿童医院遗传与出生缺陷防治中心北京市儿科研究所出生缺陷遗传学研究北京市重点实验室儿科重大疾病研究教育部重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |