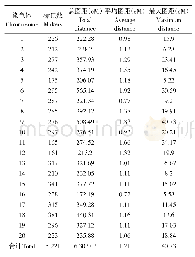
《表1 泡桐连锁遗传图谱统计》
以连锁群为单位,利用Joinmap version 4.0软件获得连锁群内标记的线性排列,多点分析估算相邻标记间的遗传距离。由表1可以看出,各个连锁群的两点标记间的平均距离变化范围是0.39~1.55 cM,连锁图的平均图距为0.58 cM。各个连锁群上标记数目变化范围是87~282个SNP,其中标记数量最多的连锁群是LG1,标记数量最少的连锁群是LG20,20个连锁群中标记密度最大的是2.57个SNP/cM,位于LG1上。在形成的20个连锁群上连锁位点覆盖的遗传距离从67.89 cM至134.49 cM,平均为102.54 cM,连锁群最长的是LG20,最短的是LG19。每一连锁群上含有的SNP连锁标记数从最少87个到最多282个。标记间平均间距最大的连锁群为LG18,平均间距为1.50 cM。从构建的2个亲本遗传图谱连锁群的数量来看,F1代连锁图包含了20个连锁群。
| 图表编号 | XD0085445600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.09.01 |
| 作者 | 李文杨、王娟、赵振利、范国强 |
| 绘制单位 | 信阳农林学院林学院、河南农业大学泡桐研究所、河南农业大学泡桐研究所、河南农业大学泡桐研究所 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式