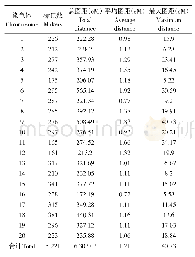
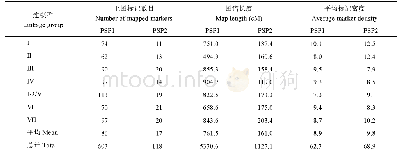
《表1 几种常见食用菌的基因组和遗传连锁图谱信息》
选择合适的分子标记是构建高质量遗传连锁图谱的关键,理想的分子标记具有高多态性、共显性、分布均匀、检测手段简单、重复性好等特点。随着分子标记技术的发展,食用菌遗传连锁图谱的研究取得了长足进步,多种分子标记在食用菌遗传连锁图谱的构建中得以应用。限制性内切酶片段长度多态性(Restriction Fragment Length Polymorphism,RFLP)标记用于构建糙皮侧耳Pleurotus ostreatus的遗传连锁图谱[10];限制性片段长度扩增多态性(Amplified Fragment Length Polymorphism,AFLP)标记和酶切扩增多态性序列(Cleaved Amplified Polymorphic Sequences,CAPS)标记用于构建双孢蘑菇的遗传连锁图谱[11];刺芹侧耳Pleurotus eryngii遗传连锁图谱构建也同样应用了AFLP标记[12];序列相关扩增多态性(Sequence-related amplified polymorphism,SRAP)标记和目标区域扩增多态性(Target Region Amplification Polymorphism,TRAP)标记用于构建香菇Lentinula edodes的遗传连锁图谱[13];特定序列扩增(Sequence Characterized Amplified Regions,SCAR)标记用于构建金针菇Flammulina velutipes的遗传连锁图谱[14]。虽然这些分子标记已被广泛并有效地应用于遗传图谱的构建,但存在重复性差(如RFLP,AFLP,SRAP和TRAP)、数量限制(如CAPS)、标记片段在基因组中分布不均匀、难以准确和完整地提供目标QTL(Quantitative Trait Loci)的相关信息等缺点。随着基因组测序和重测序技术的迅速发展,出现了新型分子标记SNP(Single Nucleotide Polymorphism),该标记在基因组中数量多、分布广,适于快速、规模化筛查,是目前QTL精确定位的首选标记之一[15]。Gao等利用SNP标记分别构建了2个不同双孢蘑菇群体的遗传连锁图谱,成功定位了机械损伤敏感性和菌盖颜色性状的QTL[16],利用简化基因组测序技术开发SNP标记构建了白灵侧耳Pleurotus tuoliensis的高密度遗传连锁图谱[17]。高密度遗传连锁图谱可完善食用菌基因组组装,是研究食用菌的遗传机制和分子育种的基础(表1)。
| 图表编号 | XD00113164800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.12.01 |
| 作者 | 张妍、黄晨阳、高巍 |
| 绘制单位 | 中国农业科学院农业资源与农业区划研究所、农业部农业微生物资源收集与保藏重点实验室、中国农业科学院农业资源与农业区划研究所、农业部农业微生物资源收集与保藏重点实验室、中国农业科学院农业资源与农业区划研究所、农业部农业微生物资源收集与保藏重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式