《表1 真实网络信息:一种拓扑与生物功能一致的多网络比对算法》
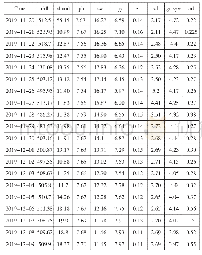
本文分别在合成网络和真实网络上测试了Con Align算法.合成网络是NAPAbench[15]中的PPI网络,这三个合成网络分别对晶体生长(crystal growth,CG)、复制-突变-互补(duplication-mutation-complementation,DM C)、带有随机突变的复制(duplication with random mutation,DMR)这三种类型的8-w ay网络进行了建模.CG中8个网络均由1000个节点和3985条边组成;DM C中8个网络的节点均是1000,边的数量分别是1919、1853、1923、1840、1867、1848、1818、1867;DMR中8个网络的节点均是1000,边的数量分别是2031、2092、1967、1977、1959、1998、2030、2056.5个真实网络分别是蠕虫(Caenorhabditis Elegans,CE)、蝇类(Drosophila Melanogaster,DM)、人类(Homo Sapiens,HS)、老鼠(M us M usculus,M M)和酵母(Saccharomyces Cerevisiae,SC).真实网络的所有数据从Iso Base[16]数据库获得,网络的节点和边信息如表1所示.生物相似性使用BLAST++工具计算得出蛋白质之间的序列相似性得分.
| 图表编号 | XD00227466000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.10.01 |
| 作者 | 夏金芳、陈璟 |
| 绘制单位 | 江南大学人工智能与计算机学院、江南大学人工智能与计算机学院、江南大学物联网技术应用教育部工程研究中心 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式