《表1 不同混合线性模型(MLM)的性能比较》
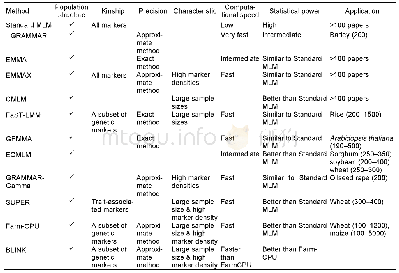
标准线性模型EMMA、EMMAX和CMLM被成百上千篇文章引用,此处省略物种及群体大小的详细统计分析。最后一列显示GWAS不同模型所研究物种和群体规模。
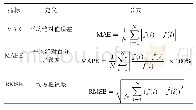
GWAS中质量性状关联分析通常采用Logistic回归模型;数量性状关联分析可以采用一般线性模型(general linear model,GLM)和混合线性模型(mixed linear model,MLM)。一般线性模型以群体结构矩阵Q或主成分分析矩阵为协变量来提高计算精度;混合线性模型利用群体结构矩阵Q、亲缘关系矩阵(kinship,K)或联合利用主成分分析矩阵和亲缘关系矩阵为协变量来抑制假关联的出现(Yu et al.,2006;Yang et al.,2014)。针对数量性状易受多因素影响的特征,混合线性模型广泛应用于数量性状的关联分析。基于混合线性模型衍生出众多方法(表1)。
| 图表编号 | XD00217995300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.11.10 |
| 作者 | 赵宇慧、李秀秀、陈倬、鲁宏伟、刘羽诚、张志方、梁承志 |
| 绘制单位 | 中国科学院遗传与发育生物学研究所、中国科学院遗传与发育生物学研究所、中国科学院大学、中国科学院遗传与发育生物学研究所、中国科学院大学、中国科学院遗传与发育生物学研究所、中国科学院大学、中国科学院遗传与发育生物学研究所、中国科学院大学、中国科学院遗传与发育生物学研究所、中国科学院大学、中国科学院遗传与发育生物学研究所、中国科学院大学 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式