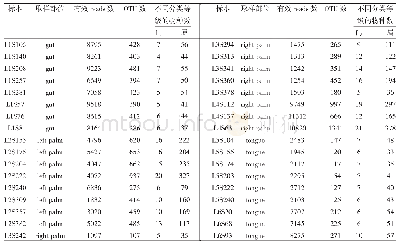
《表1 样本测序序列与OTU聚类情况统计表》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于细菌16S rRNA基因扩增子测序数据的系统发生树图制作方法》
本示例数据来自人体的肠道(gut)、左侧手掌(left palm)、右侧手掌(right palm)、和舌部(tongue),为了分析方便,省略了取样时间、抗生素使用情况等其它分组信息。通过封闭参考(closed reference)聚类法将有效测序序列按照≥97%的相似性归为一个OTU。采用RDP算法与Greengene 16S r RNA数据库比对,并将各OTU注释到所属的分类单元。使用Greengene数据库可以将大多数OTU序列注释到“属”水平,少部分OTU可以鉴定到“种”水平。通过上述方法将示例数据中的共177882条序列聚类成4403个OTU,在“门”和“属”两个水平上对同类的OTU进行分类鉴定,分别归属于24个门,615个属,有关样本的人体部位分组情况、OTU聚类和种属鉴定的具体情况见表1。OTUs聚类及分类鉴定结果与同时生成的系统发生树文件,可以作为后续统计分析,构建系统发生树图的基础。
| 图表编号 | XD00214128500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2021.01.01 |
| 作者 | 丁可、阎星羽、廉振颖、成丽娟、刁玉涛、王家林 |
| 绘制单位 | 山东第一医科大学、山东第一医科大学基础医学院、基础医学研究所、山东第一医科大学基础医学院、基础医学研究所、山东中医药大学第二附属医院、山东第一医科大学基础医学院、基础医学研究所、山东第一医科大学附属肿瘤医院 |
| 更多格式 | 高清、无水印(增值服务) |





