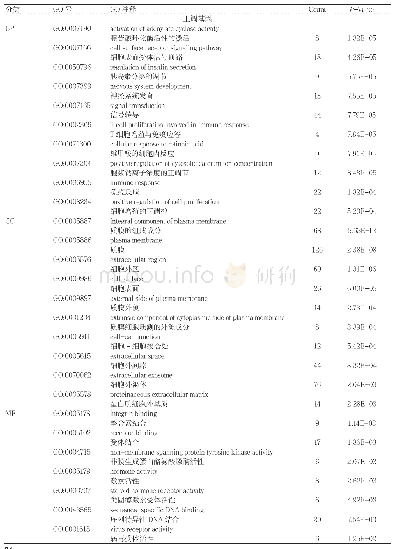
《表1 基因功能注释软件及参数》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于高通量测序的黄姑鱼转录组从头组装和基因注释分析》
使用Trinity 2.4.0软件[15]对Clean Reads进行从头组装获取转录本序列(Transcripts),所使用的参数为:--min_kmer_cov:3。基于map到转录本序列上的reads数及表达模式,Corset 1.05软件[16]进一步对获取的转录本(Transcripts)进行层次聚类,从而获取最长的聚类序列(Unigenes),软件参数使用默认参数。Unigenes序列用于后续分析。而后,利用Diamond和Blast等比对软件基于七大基因注释数据库Nr(Non-Redun-dant Protein Sequence Database)、Nt(Nucleotide Sequence Database)、Pfam(Pfam protein families database)、KOG(Clusters of Orthologous Groups of proteins)、Swiss-prot,KEGG(Kyoto Encyclopedia of Genes and Genomes)和GO(Gene Ontology)对所有的Unigene进行比对和注释,从而获得黄姑鱼的表达基因功能,基因功能注释软件及参数设置如表1。Diamond是一个用于比对数据库蛋白,其蛋白质比对性能为blast+的500~20000倍,因此比对Nr、KOG和Swiss-prot等3个蛋白数据库采用Diamond软件。
| 图表编号 | XD00168853000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.11.01 |
| 作者 | 王余菊、娄方瑞、水柏年 |
| 绘制单位 | 浙江海洋大学水产学院、浙江海洋大学水产学院、中国海洋大学水产学院、浙江海洋大学水产学院 |
| 更多格式 | 高清、无水印(增值服务) |