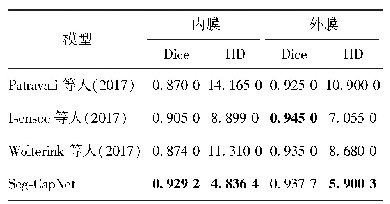
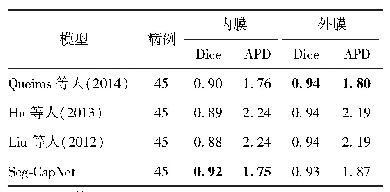
《表2 单Dice函数和多Dice函数的分割结果Tab.2 Segmentation results of single-Dice function and Multi-Dice function》下
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于三维全卷积DenseNet的脑胶质瘤MRI分割》
在本研究的初期,我们发现坏死区域和非增强肿瘤区是以往单损失函数结构最难分割的区域,但这两个区域并不是脑胶质瘤的直接分割目标,这同样限制脑胶质瘤多区域分割精度的提升。为了直接有效地分割目标区域,我们引入多Dice损失函数。如表2所示,对比原始的Dice损失函数,整个肿瘤区域的分割精度与后者几乎没差别。这可能是由于整个肿瘤区是所有除背景以外的组织区域合集,虽然单Dice结构需要同时学习各种组织之间的差异,但是背景的差异与肿瘤内组织的差异已经足够明显,所以多Dice损失函数并没有提升整个肿瘤的分割精度。增强肿瘤由于采用造影剂提升与其他组织的对比度,使得两者的差异相对明显,因而Dice系数提升较不显著。肿瘤核心区是由增强肿瘤、坏死组织和非增强肿瘤组成,相比整个肿瘤区的构成缺少水肿组织。水肿组织和肿瘤组织之间的边界往往较不清晰,导致单Dice损失函数较难同时学习好两者之间的差。我们提出的多Dice损失函数对于每一类区域都有一个分支进行针对性地特征提取,所以能显著地提升该区域的分割精度。Andrew[14]提出类似的想法,采用多个级联的三维U-Net分步预测脑胶质瘤各个区域的轮廓。为了将大区域的分割结果作为小区域的输入,该结构必须逐步训练,使得网络训练时间大幅增加。本网络结构同时输出多个区域的分割结果,有效减少网络的训练时间。
| 图表编号 | XD0016635700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2018.06.20 |
| 作者 | 黄奕晖、冯前进 |
| 绘制单位 | 广东省医学图像处理重点实验室、广东省医学图像处理重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |