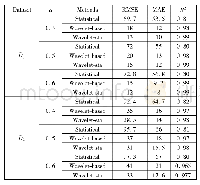
《表3 deepScore-α、Comet和MSGF+在ProteomeTools2中打分结果比较》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于多头注意力机制和残差神经网络的肽谱匹配打分算法》
打分算法的泛化性能是十分重要的,好的泛化性能能极大地提高打分算法在不同数据集中的表现而不只是在特定某一数据集下表现良好。本文首先在Humanbody数据集上训练模型,再使用训练完成的模型对ProteomeTools2中随机抽取的谱图进行打分并与Comet和MSGF+进行比较以评估其泛化性能。同样先使用Comet和MSGF+进行鉴定,再使用deepScore-α对二者输出的前50条鉴定结果进行重新打分和排序,FDR曲线如图8和图9所示。通过分析比较发现,deepScore-α在FDR=0.01条件下保留的肽序列相较于Comet和MSGF+最高提升了约7%,在30碰撞能量下完全优于Comet,在35碰撞能量下存在部分与Comet基本持平的情况,与在Humanbody数据集中进行打分的表现一致,应该为35碰撞能量数据量相较30碰撞能量不足所致。综合来看,deepScore-α具有较好的泛化性能,利用Humabody数据集训练的模型最终在ProteomeTools2上的打分效果基本与原数据集上的一致,都优于Comet和MSGF+。同样的,打分算法的Top1的命中率也是其性能评价的一个重要指标。本文也评估了deepScore-α在ProteomeTools2数据上的Top1命中率,实验结果如表3所示,deepScore-α在ProteomeTools2数据集上的Top1命中率相较于另外两个鉴定工具均提高了5个百分点,Top1的鉴定结果中Decoy的数量与另外两个鉴定工具相比减少了60%左右,因此可以判断deepScore-α在打分性能上优于Comet和MSGF+,且具有较好的泛化性能。
| 图表编号 | XD00163196300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.06.10 |
| 作者 | 闵鑫、王海鹏、牟长宁 |
| 绘制单位 | 山东理工大学计算机科学与技术学院、山东理工大学计算机科学与技术学院、山东理工大学计算机科学与技术学院 |
| 更多格式 | 高清、无水印(增值服务) |