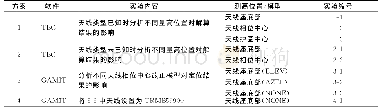
《表4 PEAKS与Spectronaut软件处理DIA数据性能比较》
对同一批DIA数据利用PEAKS Studio10.0(Bioinfomatics Solutions Inc.)和Spectronaut 11.0(Biognosys AG 2013)两种搜索工具分别进行分析,从软件对服务器的要求、处理步骤和效率、定性定量结果等方面比较两种软件的性能。从表4分析结果可以看出,两种软件在分析性能方面各有优缺点。PEAKS可通过直接搜库的方法对DIA数据进行处理,因此操作步骤较为简单,省去构建DDA实体库的步骤,前期准备工作较少;然而由于其融合de novo的算法对肽段进行定性分析,在分析时间上要远长于SPE的数据处理时间。同时,PEAKS软件定量的肽段数目有23 891,高于SPE软件的14 641,但是蛋白定量数目明显低于SPE软件,说明de novo的方法对肽段的鉴定数量有了较大的提升,然而对蛋白的定量结果并没有明显帮助。分析其中原因,可能是由于DIA数据较为复杂,直接进行线上搜库匹配的方法使得谱图匹配效率降低。SPE通过构建实体库的方法进行DIA数据分析,大大降低了谱图解析难度,增强了分析效果。除此之外,PEAKS是利用一级谱图母离子峰面积或者峰强度对蛋白定量,为了达到定量要求,一级谱图信噪比要求较高,增大了低丰度蛋白的定量难度,导致很多低丰度不能被有效定量[22];而SPE在二级谱图的基础上根据子离子峰面积或者峰强度进行定量,有利于信噪比的提高和低丰度蛋白的检出,增强了定量能力[8]。
| 图表编号 | XD00156698600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.08.20 |
| 作者 | 窦玥、熊为亮、梁如冰、侯敬丽 |
| 绘制单位 | 上海交通大学药学院、上海交通大学生命科学技术学院、上海交通大学生命科学技术学院、上海交通大学分析测试中心 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式