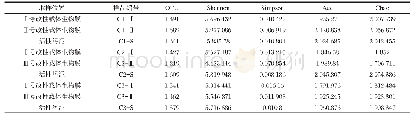
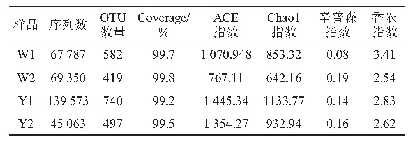
《表2 样品OTU及多样性指数》
利用16S rRNA基因高通量测序技术对阳宗海南岸湖滨带底泥细菌进行基因测序,共得到有效序列187 056条(表2)。利用UPARSE软件[15]对测序样品的有效条带按照97%的一致性进行聚类为OTU。虽然16S rRNA基因高通量测序技术已被广泛用于微生物生态学研究中,但鉴于16S rRNA基因内部基因组存在异质性,会导致过高估计微生物多样性的情况,很少有研究考虑了基因组内异质性的影响[16]。该研究选择V3-V4区间,作为细菌目标靶向,而V4区域作为基因组内异质性最小的区域,被认为是细菌理想的靶标,从而确保微生物数据的准确性,进而对阳宗海南岸底泥微生物多样性进行正确的计算。
| 图表编号 | XD00124790600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.02.20 |
| 作者 | 赵立君、刘云根、王妍、赵蓉、任伟、徐鸣洲 |
| 绘制单位 | 西南林业大学湿地学院、西南林业大学生态与环境学院、西南林业大学生态与环境学院、西南林业大学生态与环境学院、西南林业大学生态与环境学院、西南林业大学生态与环境学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式