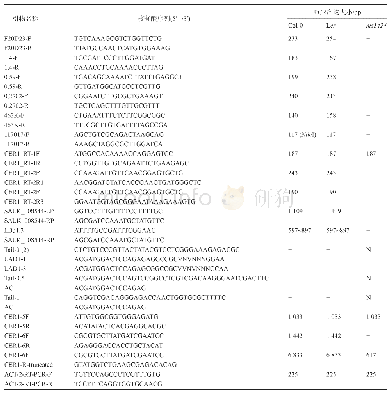
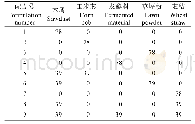
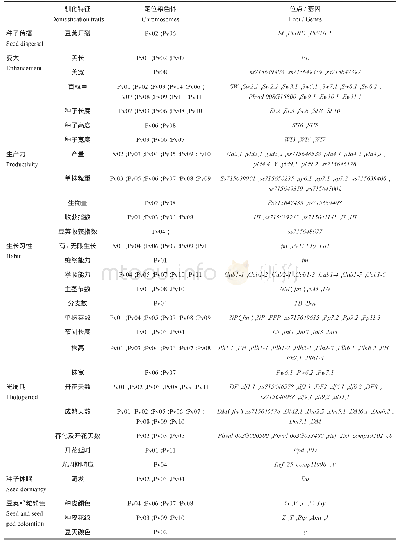
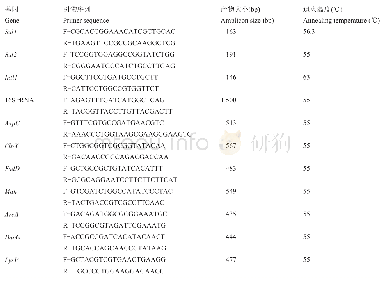
《表2 主要作物驯化基因组位点的定位与鉴定》
目前,对作物驯化基因组区段的解析研究主要通过基于基因组重测序的群体遗传学分析途径和基于连锁遗传理论的数量性状位点研究手段开展,近年来取得的突破性进展主要集中在水稻、玉米、大豆、菜豆等作物中(表2)。其中,二代基因组测序技术结合核心种质群体的群体遗传分析已经成为主流,代表性的进展如:Huang等[29]采用二代基因组重测序手段,通过系统解析野生稻群体与栽培稻群体的全基因组核酸多样性的变异情况,解析出了55个水稻受到驯化选择的基因组区段,并且通过分析发现,水稻已知驯化相关基因如BH4(种皮色)、SH4(落粒性)、PROG1(匍匐生长习性)、qSW5(种子宽度)、OsC1(叶鞘颜色)等均位于这些驯化相关基因组区段内,该研究从侧面证实了采用群体遗传分析手段解析作物驯化相关遗传区段的准确性、有效性和必要性;Hufford等[30]采用二代基因组重测序技术,解析出了484个与玉米驯化紧密相关的基因组区段,这些基因组区段占到玉米基因组的7.6%,平均长度为322 kb,平均涵盖3.4个注释基因,结果显示,玉米驯化过程中,存在除了TB1(分支分蘖性)、TGA1(坚硬外稃)等已知基因之外的更多的基因,有待进行深入的解析和研究,为深入认识和分析玉米的驯化过程提供了候选基因组区段;Zhou等[31]采用二代基因组重测序技术,使用XP-CLR算法,分析出了121个与大豆驯化直接相关的基因组位点,并且发现这些驯化基因组位点与多个已知驯化基因位置重合,得到的驯化区段定位精度远远超过传统QTL分析方法,具有更高的解析效率。该研究同时采用了全基因组关联分析的手段,发现了多个控制大豆籽粒油分含量,籽粒重量及茎秆建成紧密相关的信号位点受到了驯化选择,为认识大豆的驯化过程提供了候选基因组区段;Schmutz等[32]采用二代基因组重测序技术对菜豆种质资源开展研究,利用滑框分析方法,获得了930个在野生种及栽培种间基因组多样性参数及选择分化系数显著差异的基因组区段,累积影响了超过74 Mb的菜豆基因组,约占整个菜豆基因组的八分之一。此外,还发现了209个驯化区段的平均长度超过100 kb,系统解析了普通菜豆两个独立驯化亚群的驯化区段相似度,提出了菜豆两次独立驯化的遗传证据;Li等[33]采用二代基因组测序技术对桃种质资源开展测序,采用滑框分析方法,鉴定出了142个在野生种及栽培种间基因组多样性参数极显著差异的基因组区段,累积影响了超过42.4 Mb的桃基因组,约占整个桃基因组的18.6%。通过全基因组关联分析,鉴定出了多个与桃果实大小和风味相关的遗传位点受到了驯化选择。这一研究为认识桃的驯化起源过程提供了新的遗传证据。
| 图表编号 | XD00114632600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.11.16 |
| 作者 | 贾冠清、孟强、汤沙、张仁梁 |
| 绘制单位 | 中国农业科学院作物科学研究所、中国农业科学院作物科学研究所、中国农业科学院作物科学研究所、中国农业科学院作物科学研究所 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式